- Sanger Sequencing
- dNTP : DNA polymerase가 dNTP의 상보적인 DNA를 합성
- 산소 한개가 떨어진 ribose
- ddNTP(dideoxy nucleotide) : 합성 중단, 염기에 따라 다른 형광
- 산소 두개가 떨어진 ribose
- 모든 조각들의 마지막에는 dd가 들어간다.
- 주형의 서열을 알고 싶다면 primer 를 붙여야 한다.
- dNTP : DNA polymerase가 dNTP의 상보적인 DNA를 합성
- Automated DNA Sequencing
- Sanger가 느려서 만든 것.
- Sanger와 원리가 같지만 ddNTP에 다른 형광물질을 붙여서 사용한다.
- 전기영동으로 분리하며 하단에 (+) 전하로 (-)인 DNA가 짧은 순으로 끌려간다.
- (+) 전하쪽으로 빠져 나올 때 Laser를 쏜다.
- Chain termination
- single strand DNA 의 염기서열을 상보적인 DNA로 합성할 때 특정 염기에서 반응이 중지되게 하는 방법
- Sequencing에 쓰이는 DNA polymerase
- DNA polymerase 1
- 5’ -> 3’ exo
- 3’ -> 5’ exo
- Klenow fragment
- 3’ -> 5’ exo
- Sequenase
- 3’ -> 5’ exo
- DNA polymerase 1
- Next Generation Sequencing
- Sanger와 다르게 병렬 sequencing이 가능하다.
- Roche(454)
- 하나의 bead에 하나의 DNA 사슬 고정 후 emPCR로 증폭시킨다.
- 증폭과정에서 인산에스테르 결합이 일어나면서 PPi가 방출된다.
- Luciferase가 발광하는 염기의 빛을 측정하여 읽어낸다.
- 남아있는 dNTP들은 Apyrase로 제거한다.
- Pyrosequencing 기법을 이용한다. : PPi가 나오느냐 안나오느냐로 염기가 중합되는지를 판단
- illumina / solexa
- single strand DNA 조각의 양 끝에 adaptor를 붙인다.
- Flowcell 바닥에는 adaptor 서열의 상보적인 DNA를 고정시켜 놓고 adaptor가 달라 붙게 한다.
- 양 끝이 결합되면 Bridge를 만들고 생성된 이중가닥을 primer로 인식하여 PCR한다.
- Polony(Polymerase colony)가 형성되고 형광물질을 읽으면서 sequencing 한다.
- NNGS
- amplification 없이 single molecule sequencing. 증폭할 필요가 없다.
- PacBio Sequencing
- 바닥에 DNA polymerase 심고 polymerase가 sequencing 한다.
- Signal이 약할 때 사용함
- Ion Torrent Sequencing
- 한 염기씩 추가될 때 수소이온(H+)이 발생하고, pH의 미묘한 변화를 감지하며 염기서열로 인식
- Oxford nanopore sequencing
- Nanopore에 DNA 단일 가닥이 통과될 때, 염기서열에 따라 막 간의 전위차를 통해 염기서열 해독
- Nanopre 밖에서 helicase가 DNA를 풀면서 단일 가닥이 통과된다.
- Genome Sequencing 방법
- Shotgun approach
- DNA를 잘게 토막낸 후 염기서열 분석
- 서로 겹치는 부분을 컴퓨터가 찾아내어 순서를 짜 맞춘다.
- Clone contig apporach
- Chromosome walking : 클론의 말단 부위를 가지고 중첩되는 Contig를 작성하는 것
- Clone fingerprinting : 특이한 반복서열 찾기
- Moleular markers : EST, STS, STR, SNP 이용
- Shotgun approach
10장 - Next Generation Sequencing
목차
1. Sanger Sequencing
2. Automated DNA sequencing
3. Chain termination
4. Next Generation Sequencing (NGS)
- Roche(454)
- Illumina/Solexa
5. Next Next Generation Sequencing (NNGS)
Sanger Sequencing

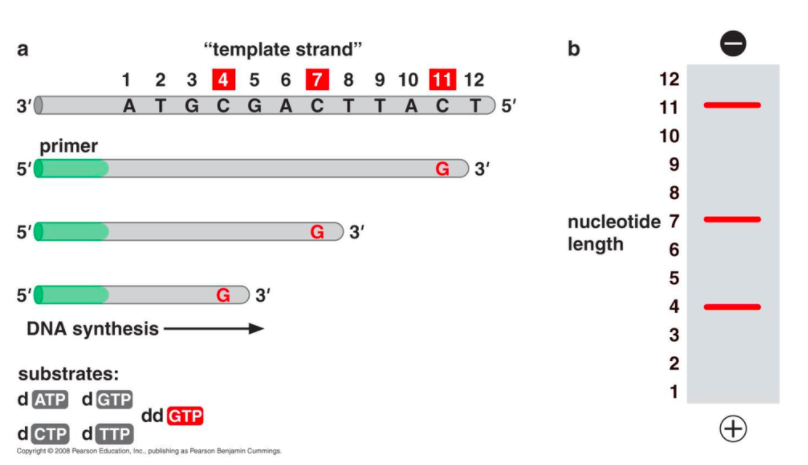
1977년 생거 등에 의해 개발된 염기서열 분석법으로서 1980년 생거가 두번째 노벨상을 받게된 계기다. 위의 그림 상단에 나와있는 3'-ATGACTGAGC-5'와 같은 template DNA(주형DNA)의 서열을 분석하는 경우에, 일반적인 d{A,C,G,T}TP (NTP)만 넣어주면 DNA polymerase는 이에 상보적인 DNA를 합성하게 된다. 하지만 dNTP 외에 소량의 dd{A,C,G,T}TP (ddNTP)를 섞어주게 되면, DNA polymerase가 template DNA에 상보적인 서열을 합성해 나가다가, 중간중간에 ddNTP가 끼어 들어간 DNA 분자가 합성되게 된다. 그러한 분자는 더 이상 길어지지 않고 합성이 중단되게 된다. 이들 ddNTP에는 각각을 구별할 수 있는 형광물질이 결합되어 있기에, 새로이 합성된 DNA 들의 마지막 염기 종류에 따라 서로 다른 형광을 띄게 된다. 여기에서 주목할 것은, 합성된 DNA 분자들은 모든 자리에서 4개 중의 어느 하나의 ddNTP가 끼어 들어간 것이기에, 정확히 한 염기씩 길이의 차이가 나는 것들이다. 이들은 전기영동법에 의해 크기 순으로 나열할 수 있으며, 레이저 빛을 각 전기영동밴드에 쬐면, 형광물질에 따라 특이적인 파장의 빛을 발하게 되며, 이를 순서대로 읽으면 원래 염기서열과 상보적인 5'-TACTGACTCG-3'을 얻게 된다.
1. NTP는 Nucleotide Tri-Phosphate의 약자이다.
DNA를 구성하는 염기를 우리는 아데닌 구아닌 시토신 티민 등등으로 표현하는데, 이것은 단지 거기에 붙어있는 염기 물질의 이름만을 따서부르는 것이다. 실제적으로는 nucleotide 라고 하는 구조로 되어 있다. 이것은 기본적으로 인산 - 당 (5탄당인 ribose) - 염기로 구성됩니다. 인산이 하나 붙으면 nucleotide mono-phosphate (NMP), 인산이 두개 붙으면 nucleotide di-phosphate (NDP), 3개 붙으면 nucleotide tri-phosphate (NTP)라고 부릅니다.
2. dNTP와 ddNTP는 각각 deoxy-nucleotide tri-phosphate와 dideoxy-nucleotide tri-phosphate의 약자이다.
nucleotide의 구조 중에서 중간에 당이 들어간다. 5탄당 (탄소 5개로 구성되었다는 뜻)의 ribose가 이용되는데, 여기서 산소가 하나 떨어진 상태의 ribose가 포함된경우에 dNTP, 두개가 떨어진 ribose를 가지고 있으면 ddNTP라고 한다.
정상적이지 않은 ddA ddT ddG ddC가 들어오면 멈추게 된다. 얘내들의 특징은 사이즈가 다 다르다. 한 배수씩 사이즈가 다르다. 1부터 10까지. 전기영동 gel에다가 로딩을 하고 ,DNA는 음전하고 gel은 밑이 양전하기 때문에 밑에서 땡겨주게 되면 가벼운 것이 밑으로 무거운 것이 위로 배열되게 된다.
이 때 ddA ddT ddC ddG에 서로 다른 형광물질을 labeling해서 서로다른 색깔로 구분할 수 있다.
짧은게 맨 밑 긴게 맨 위 -> 이걸 통해 sequence를 알 수 있다.

지금 막 중합시킨 것이 뭔지 확인하면서 원래 주형의 DNA서열을 결정한다.

매 염기마다 종결되게 하는 NTP가 있다.

dNTP(왼쪽)는 계속 중합이 되지만, ddNTP(오른쪽)가 들어가면 멈춘다.

3'에 중합이 된다.
Dideoxy Nucleotide가 들어가면 거기서 끝난다.

template strand(주형)이 있고 주형의 서열을 알고 싶다면 프라이머(primer)를 붙여야 한다.
사이즈 마커를 넣으면 몇번째가 들어갔는지 확인할 수 있다.

비정상적인 A 나 T G C를 넣음으로써 뭘로 끝났는지 앎으로써 DNA 서열을 알 수 있다.

모든 조각들의 마지막은 dd 가 들어간다.
A dT dT dG dC … ddT
...
A T T G ddC
A T T ddG
A T ddT
A ddT
ddA
Automated DNA Sequencing

1세대 기법은 자동화 기법이다. Sanger는 0세대

Color 코드를 읽는 것이다.


왼쪽은 Sanguer Sequencing으로 나온 서열, 오른쪽은 자동화 기법이다.




전기영동으로 분리되는 과정이다.
DNA는 (-) 전하를 띄기 때문에, 전기영동 장치의 하단에 있는 (+) 전하로 짧은 순으로 끌려간다.





(+) 전하쪽으로 빠져 나올 때 Laser를 쏘면
각 종류별 형광물질이 나오게 되면서, 기본적인 Sanger Sequencing이 완성된다.

Chain termination
Chain termination method : single strand DNA 분자의 염기서열을 상보적인 DNA의 효소적 합성할 때 특정 염기에서 반응이 중지되게 하는 방법
(1) single stranded DNA 가닥을 만듦
(2) single stranded DNA 에 oligonucleotide(합성한 것)을 annealing 하여 primer로 사용하게 함
(3) 4종의 dNTP와 1종의 ddNTP를 넣고 중합효소로 중합 (첨가한 ddNTP에 따라 4가지 반응)
(4) 전기영동

Chain termination 이 왜 일어나는지를 보여준다.



먼저있던 가닥의 5’을 지울 수 있다. (a)
시퀀싱에서는 5’ to 3’ activity는 좋지 않다. 임의로 크기를 지우기 때문에.

2. Kelnow fragment는 일부를 잘라냈기 때문에 processivity가 좀 낮다.
3. Sequenase 5’to3’ 없고 길게 만들수 있고 3’->5’은 가지고 있다.
5. Sequenase .

PCR을 하는데 똑같이 ddA를 넣는 것이다.


정상적인 G가 들어가면 계속 가지면 비정상적인 G가 들어가면 멈추게 된다.
반응이 다 끝난 다음에 전기영동을 하게 된다.
0세대 시퀀싱



ddA ddC ddT ddG 동위원소로 표지한다

Next Generation Sequencing (NGS)
Sanger sequencing method는 많이 개선되어 현재는 완전히 자동화 되었다. 하지만 하나의 테스트 튜브에서는 한 조각의 DNA만 시퀀싱할 수 있다. 한 종의 게놈을 시퀀싱하기 위해서는 게놈을 마구잡이로 조각을 내어 각각을 시퀀싱하고 이들을 컴퓨터 알고리즘으로 조립하는 방식을 취하기 때문에, 박테리아 게놈을 생거방법으로 시퀀싱하기 위해서는 수만개의 조각을 각기 독립적으로 시퀀싱해야 한다. 한 대의 시퀀싱 기기에서는 한번에 수천개 정도의 조각만을 시퀀싱 할 수 있다. 서열을 따라가면서 한 bp씩 차이가 나는 DNA 분자를 합성하고 이를 전기영동으로 분리한 후에 형광으로 검출하기 때문에, 많은 양의 시약이 소모되게 된다. 따라서 동시에 시퀀싱할 수 있는 DNA 조각의 갯수가 제한적이고, 각 조각을 시퀀싱하기 위해서 많은 양의 시약이 소모되는 생거 시퀀싱의 한계를 극복할 수 있는 방법이 Next Generation Sequencing(NGS)이다.

NGS의 과정
1) 샘플 준비 : 조직, 혈액 등에서 genomic DNA 또는 RNA 추출
2) 라이브러리 제작 : DNA를 조각으로 자르고, 라이브러리를 제작한 뒤, 양과 질을 확인
3) 클러스터 제작 : 주형을 증폭시킴
– 오일과 물의 성질을 이용한 에멀전을 이용한 PCR법 (emPCR)
: 454, SOLiD, Ion Torrent, GeneReader)
– 다리 형태로 증폭되는 PCR 법(Bridge amplification) : Illumina
4) 시퀀싱 : 증폭된 주형의 염기 서열을 분석
– probe의 연결에 의한 시퀀싱(sequencing-by-ligation) : SOLiD, Complete Genomics
- 염기가 주형에 결합하면서 시퀀싱 (sequencing-by-synthesis)
: Illumina, GeneReader, 454, Ion Torrent
5) 데이터 분석

기본적으로 모든 시퀀서는 Sanger 방식 기반이었는데, 최근 새로운 기법 의 시퀀싱 기술이 급속도로 보급되었으며, Sanger 방식과 달리 대량의 병렬 데이터 생산이 가능한 이러한 시퀀서를 차세대 또는 제2세대 시퀀서 (Next Generation Sequencer, 2nd Generation Sequencer)라고 한다. NGS 의 등장은 이제 무어의 법칙마저 능가하는 속도로 게놈 시퀀싱 비용을 감소시키고 있으며 곧 인간 한 사람의 게놈 시퀀싱이 $1,000 달러에 가능하게 되리라는 것이 명확해 지고 있다

세가지 주된 NGS 방법이 있다.
- Roche(454)
- illumina/solexa
- SOLiD
NGS에서는 기종에 따라 약간의 차이는 있지만 기본적으로 DNA 서열에 대한 증폭을 하고, 그 후 형광표식 등을 카메라로 찍어 이미지 처리를 하는 과정을 거쳐 염기를 읽어낸다. PCR 증폭 방식에 따라서 로슈 (Roche, 454)와 라이프 테크놀로지스 (Life Technologies)의 기종들은 emulsion PCR (emPCR) 방식으로, 일루미나 (Illumina)의 기종들은 solid-phase amplification을 사용하는 것으로 나눌 수 있다.
1. Roche(454)
Roche(454) 장비의 원리
- 454 시리즈에서는 하나의 bead에 하나의 DNA 사슬을 고정한 후에 emPCR로 증폭을 시킨다.
- 친수성과 소수성 성질을 이용하여 오일로 된 emulsion 기포를 만들고, 기포 안에서 한 가지의 adater를 primer로 해서 DNA가 증폭될 수 있도록 bead와 증폭에 필요한 시약들이 포함되도록 한다.
- 증폭이 끝나면 각 bead에는 수백만 개로 복제된 동일한 DNA 서열이 덮이게 되고, 각 bead는 PicoTiterPlate라는 장치에서 하나의 구멍(well)에 하나씩 들어가서 시퀀싱에 개시된다.
- 증폭 과정에서 염기가 결합하면서 발생하는 2개의 phosphate이 ATP로 변하며 PPi가 방출되고
- 이루어지는 일련의 과정을 통해 oxy luciferin이 생성되면서 화학적 발광 (chemiluminecence)을 띄게 되는데 이 빛의 신호로 염기 서열을 확인할 수 있다.
- 454의 시퀀싱 기법은 Pyrosequencing라고 하며 Luciferase가 사용되어 염기의 빛을 측정하여 읽어낸다.

염기를 하나씩 차례대로 뿌린다. dCTP를 뿌렸다면 중합이 된다
인산이 결합이 될때 PPi 가 나오고 ATP가 만들어지고 Luciferase가 발광하며 빛이 나온다.
뿌리고 나서 여분으로 남아있는 dNTP들은 지워야한다.
이때 Apyrase로 지운다 ****
이게 pyrosequencing이다 : PPi가 나오느냐 안나오느냐로 염기가 중합되는지를 판단하기 때문
이게 Roche454에서 쓰는 방법이다.

G뿌리고 중합됐다. C뿌리고 중합됐다. T뿌렸더니 중합 안됐다.
중합이 된 서열은 PPi를 검출함으로써 알 수 있다.

Bid에 붙인다.
A를 쫙 뿌리면 빛이 나오는 구멍이 있고 안 나오는 구멍도 있다.
합성하고 이미지 체크하고, 또 한번 합성하고 이미지 체크하고 하면서 진행한다.

Sequencing by Synthesis : 실기간 합성하고 염기가 뭔지 알아내는 것

판 위에 붙이는(bridge PCR) 시퀀싱 방법이 있고, Bid에다가 붙이는(emulsion PCR) 시퀀싱 방법이 있다.
Illumina / solexa
Illumina/Solexa 장비의 원리는 DNA를 단편으로 자른 뒤, 양쪽에 다른 종류의 adaptor를 붙여 만든 라이브러리를 이용한다. 제작된 라이브러리를 adaptor와 상보적인 oligo가 붙어있는 Flow cell 이라는 슬라이드에 흘려 보내어 증폭시킨 뒤, DNA를 합성시킬 때 염기가 끼어들면서 나타나는 형광을 측정하는 Sequencing by synthesis 기술을 이용하여 염기서열을 분석하게 된다.
일루미나의 NGS 기법에서는 브릿지 증폭이라고 해서 슬라이드 위에 DNA 단편을 고정시킨 후에 최대 1,000 분자까지 증폭시켜 같은 서열의 DNA 단편 집단을 형성시키는 게 특징인데, 일루미나에서는 이 집단을 클러스터 (cluster)라고 표현하며, 이 클러스터를 주형으로 네 종류 의 형광 표식 염기를 사용한 일 염기 합성반응인 SBS를 수행한다.
장점은 염기 서열 분석 시 단가를 획기적으로 줄였다는 것이고, 단점은 한번에 읽을 수 있는 염기 서열의 길이가 상대적으로 짧다는 것이다.

- single-strand DNA Fragment들을 준비한다.
- 우리가 시퀀싱 하고자 하는 서로 다른 수십억 개의 single-strand DNA 조각들의 양쪽 끝에 double-stranded linker(adaptor)를 붙여준다.(linker나 adapter라고불리우는 것을 붙여준다.)
- Flowcell 바닥에는 이들 adaptor 서열에 상보적인 DNA를 고정하여 놓고, adaptor가 붙어있는 조각들을 flowcell에 고루 퍼지게 하면, 각 조각은 자기 파트너에가서 달라붙게 된다.
- 양끝이 모두 결합되면 Bridge처럼된다. 이때 생성된 이중 가닥 부분을 primer로 인식하여
- Polymerase chain reaction(PCR)을 하여 증폭 시킨다.
- PCR이 진행되면 새로 합성된 DNA 가닥들은 바닥에 고정되고 고정된 DNA 가닥은 반대 방향 adapter에 의해 primer와 결합하여 다시 합성된다. (다시 풀게되면 원래거랑 상보적인 것이 만들어지는 것을 계속~~)
- 이 과정이 반복되면서 증폭이 끝날 때는 하나의 DNA 조각에 약 1000개 정도의 동일한 PCR 결과물이 하나의 cluster를 이루게 된다. 즉, 애초에 사용한 DNA 조각하나로부터 하나의 클러스터가 형성되며, 이들은 다음 단계에서 사용하는 형광법으로 검출하기 충분한 양이 되는 것이다.
- 이때, 한쪽 가닥은 없애고 나머지 가닥만을 시퀀싱한다.
- 이 cluster들에 염기의 종류에 따라 각각 다른 빛을 내는 형광 물질이 결합된 dNTP가 DNA polymerase에 의해 한 염기씩 합성될 때, 염기마다 형광 물질 빛을 읽은 뒤 기록한다. 기록한 형광 물질은 washing으로 제거한 뒤 새로운 dNTP가 합성되면 위 과정을 반복한다. 여기서 중요한 것은, 한 염기씩 붙이고 형광을 검출한 후에, 그형광을 없애고 그 다음 염기를 붙여야 한다는 점이며, 이 과정이 착오없이 차근차근 진행되어야 한다는 점이다.(형광을 차근차근 떼어 내야 그 다음 반응이 이루어진다.) 이 cycle은 약 100번 정도 반복되며, 결과적으로 수십 억 개의 DNA 조각들에 대한 약 100bp 정도의 sequence를 얻을 수 있다. 이러한 방법을 sequencing by synthesis라고 부른다. 정해진 염기 수만큼 시퀀싱이 끝난 후에, 시퀀싱한 조각에 상보적인 가닥을 합성하여 시퀀싱함으로써 결과적으로 앞, 뒤 서열을 얻을 수 있는데 이를 “Paired-end sequencing”이라고 한다

DNA 칩 위에 뿌린다.
빨간 거에 상보적인 것도 있고 파란 거에 상보적인 것도 있다.
Polymerase colony -> Polony라고 부른다.
Polony가 형성되면 4가지 칼라코드 된 애를 쫙 뿌린다.
그 스팟에 무슨 색이 들어갔는지 detect한다.
b에서 하나씩 뿌리면서, 합성하면서 한개 한개 서열이 결정된다.

Genome을 조각내고
adaptor를 붙이고
adpator 짧은조각의 프라이머가 붙어있는 곳에 뿌리면
각 스팟의 조각들이 분산이 된다.
상보적인 조각을 뿌려놨기 때문에 이중가닥을 형성하면서 붙어있게 된다.

Next Next Generation Sequencing (NNGS)

NNGS는 Signal이 약하게 나와도 얼마든지 detect할 수 있게 하기 위해서 나왔다.
따라서 증폭할 필요가 없다. 시간도 덜 걸리고 가격도 싸졌다.
NNGS 중 중요한 3가지
1. SMRT (single molecule real time) = PacBio Sequencing
하나하나 중합하면서 바닥에 DNA polymerase를 심어놓고 Polymerase로 하여금 sequencing 하게 한다..
ZMW의 표면으로 하면 약한 signal도 인식한다.
2. 반도체 Sequencing
Pyrosequencing이나 초노빨파 이런 형광이 아니라 전혀 다른 것을 감지하는데
인산에스테르 결합이 일어나면서 PPi도 형성되지만 다른 것도 형성되면서 pH의 미묘한 변화를 감지하면서 염기서열로 인식한다.
식중독의 원인균을 최초로 해독을 했다.
3. Oxford nanopore sequencing
아주 미묘한 전류변화의 차이를 통해 염기서열을 해독한다.
중합이 되지 않아도, 그냥 통과할 때도 전류변화의 미묘한 차이를 통해서 해독한다.
Single Molecule Real Time (SMRT) = PacBio Sequencing

하나 중합해놓고 지금 중합된게 뭔지? 보는 것이 Real-time sequencing 이었다.
단 한 분자만 붙어도 충분히 민감한 센서를 통해서 캐치할 수 있다.

각 염기에 서로 다른 형광물질을 매달아서(A,T,G,C 마다) 이를 검출함으로써 시퀀싱을 하는 점은 Sanger 혹은 Solexa 방법과 유사하다고 할 수 있다. 그런데, 형광을 염기쪽이 결합시키지 않고 떨어져 나올 인산쪽에 결합시켰기 때문에, 매 염기가 결합될 때마다 새로운 형광물질이 떨어져 나오게 되며 이를 검출하여 시퀀싱하게 된다. 따라서 terminator가 필요없게 되어 매우 빠른 속도로 시퀀싱이 가능하게 되며, 실제로 초당 10 염기 정도 시퀀싱이 가능하다고 알려져 있다.
이 방법에서는 한 DNA 분자를 증폭하여 동시에 시퀀싱하기는 불가능하며 단일 분자를 대상으로 시퀀싱을 진행하여야만 한다. 단일 분자에서 나오는 형광은 너무 약하기에 일반적인 방법으로는 측정하기가 곤란하다. 매우 작은 공간에 단일 DNA 분자를 가두어 놓고 고도로 집중된 레이저를 이용하여 측정하는 소위 Zero Mode Waveguide라는 기술을 사용하여 형광을 검출하고 있다. 단일 DNA 분자를 가둘 수 있는 극소 공간을 매우 많이 만들어 많은 샘플을 동시에 시퀀싱할 수 있다.
Ion Torrent Semiconductor Sequencing
Illumina의 Solexa 시퀀싱은 형광으로 검출하지만, Ion Torrent사의 방법은 한 염기씩 추가될 때마다 수소 이온이 발생하는 점에 착안하여 이를 전기적으로 측정하는 방법을 사용하였다.
매번 하나씩 중합될 때마다 3’ OH가 지금 막들어가려고 하는 뉴클레오티드의 H+를 날려버리게 된다. PPi가 아닌 H+가 방출되는 것을 착안한다.

그런데, A/T/G/C 중 어떤 염기가 추가되던 똑같은 수소 이온이 발생하기 때문에 이들을 전기적으로 구별할 수는 없다. 따라서 먼저 dATP를 반응시켜보고 수소 이온이 발생하는지 측정해 본다. 시그널이 나오지 않으면 남은 dATP를 씻어내고 다른 dNTP들을 차례로 반응시켜본다. 즉, 한 염기 당4번의 반응을 시도하는 것이다. (한마디로 A,T,G,C를 하나씩 넣어보는 것이다. 그리고 반응하면 A다,T다 등등)
형광법에서는 한 염기씩 반응시키기 위해서 terminator를 사용하였는데, 여기서는 terminator를 사용하지 않는다. 만약에 A 다음 염기도 A라면, dTTP 두 분자가 반응할 것이고, 수소 이온도 두 배만큼 더 많이 생성될 것이며, 전기적 시그널도 그만큼 클 것이기 때문에, 시그널의 크기로 연속적인 염기(homopolymer)가 존재하는지 알수 있다.

이 방법을 구현하려면, 많은 DNA 조각을 따로 따로 반응을 시키고 각각의 조각에서 얻은 전기 시그널이 서로 간섭하지 않게 하는 기술과 전기적으로 검출할 수 있을 만큼 충분한 양이 되도록 증폭하는 기술이 필요하게 된다. 수 많은 DNA 조각을 각기 하나의 구슬(bead)에 붙인 후[bead에 DNA를 코팅시킴] (하나의bead에는 수많은 같은 adaptor가 붙어있다), 각 구슬을 서로 다른 우물에 넣고 우물 안에서 PCR이 일어나도록 하여, 한 구슬의 표면은 동일한 DNA 조각으로 도배되게끔 한다.

계속 반복하면 결과적으로 bead에는 똑같은 DNA가 모인다. 우물 안에는 똑같은 DNA만 있다.


Oxford Nanopore Sequencing
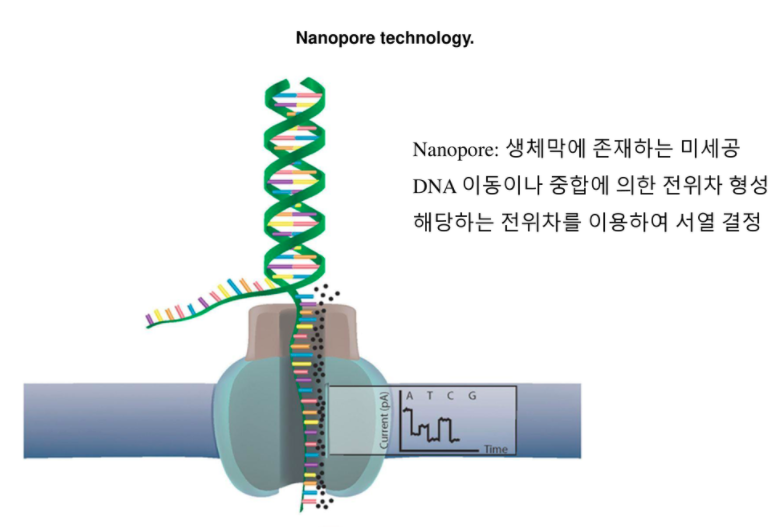
영국의 옥스포드는 막에 나노미터 수준의 채널을 형성한 후에 이곳에 DNA 단일 가닥을 통과시킬 때, 염기 서열에 따라 막 간의 전위 차가 변하는 것을 착안하여 시퀀싱을 한다. 막 바깥 쪽에서 이중 나선의 DNA를 helicase 효소를 이용하여 풀어주면서 단일 가닥이 일정한 속도로 채널 속으로 이동하도록 구성되어 있으며, 채널 안에 약 4 염기 정도가 들어가기 때문에 실제로 측정한 전위 차는 한 염기에 해당하는 것이 아니라 4 염기 단위로 얻어지는 것이다. 이렇게 얻은 데이터로부터 단일염기 서열을 복구하는 소프트웨어가 매우 중요한 역할을 한다. 이 방법의 가장 큰 장점은 다른 방법들과 달리 template DNA에 상보적인 화학 반응을 하지 않는다는점이고 DNA를 있는 그대로 측정한다는 점이며, 한번에 수만 염기를 읽을 수 있다. 따라서 특별한 기기를 필요로 하지 않고 작은 카트리지만 있으면 그 안에 실제시퀀싱이 일어나는 막을 포함한 소모품인 flowcell만 있으면 된다는 것이다. 결과는 USB 포트로 나오기에 노트북이나 스마트폰에 연결하면 되기에 휴대용이다. 채널을 집적화한 형태의 제품도 출시되었다.

Double Helix를 풀면서 Nanopore을 통과하면 전류의 세기가 달라진다.
그럼으로써 하나하나 염기를 알 수 있다.

Single Molecule sequencing , Nanopore Sequencing


각 염기에 tag를 단다.
tag가 Nanopore 통과하면서 염기서열을 알 수 있다.
Nanopore의 다양한 방법

'🧬 Bio > 유전공학' 카테고리의 다른 글
| 유전공학 11장 - Studying gene expression and function (0) | 2020.05.04 |
|---|---|
| 유전공학 10장 - Next Generation Sequencing (2) (0) | 2020.04.27 |
| 유전공학 9장 - 중합효소 연쇄 반응(PCR:The polymerase chain reaction) (0) | 2020.04.20 |
| 유전공학 8장 (2), 9장 - 유전자 및 유전체 구조 연구 (0) | 2020.04.16 |
| 유전공학 7장 (2), 8장 - 특정 유전자의 클론을 얻는 방법 (0) | 2020.04.13 |