- Designing primer
- primer가 너무 짧다면 다양한 DNA가 만들어져 background가 커진다.
- primer가 너무 길다면 주형 DNA와 혼성화를 이루는 속도가 느려진다.
- 17mer 짜리 primer가 가장 적당하다.
- Tm = (4×[G+C]) + (2×[A+T])℃
- Studying PCR product
- Agarose gel electrophoresis
- qPCR : 증폭과 정량을 동시에 하는 PCR
The polymerase chain reaction (PCR)
1970년대에 gene cloning기술의 개발은 이전에 불가능했던 방법으로 유전자와 유전자 활성에 대한 연구를 가능하게 함으로써 신선한 자극으로 받아들여졌다. 두번째 혁신적인 방법 - polymerase chain reaction(PCR) - 이 고안된 1980년대 후반에도 동일한 상황이 전개되었다. PCR method는 매우 간단한 기술이다. DNA중합효소에 의해 DNA분자의 짧은 부위가 여러번 복제되는 것이 전부다. 평범한 실험처럼 보이지만 그것은 유전학적 연구와 생물학의 더 넓은 영역에서 많이 응용되고 있다.
The polymerase chain reaction in outline
PCR은 DNA분자의 특정부위를 선택적으로 증폭시킨다. 어떤 DNA분자의 어떤 부위라도 선택될 수 있다. Border sequence들은 PCR이 수행되기 위해서 알려져 있어야 한다. 또한 두 개의 짧은 oligonucleotide가 이중나선의 각 가닥과 혼성화되어야만 한다. DNA합성반응을 위한 primer로써 역할을 하는 이들 oligonucleotide들은 증폭될 부위의 범위를 정해준다.
일반적으로 증폭반응은 Thermus aquaticus로부터 정제된 DNA polymerase I 에 의해 수행된다. 이 생물체는 온천에서 서식하고 있고, Taq polymerase를 포함하는 많은 효소들이 thermostable하다. 이것은 그들이 열처리에 의한 변성에 대해 저항성이 있다는 것을 의미한다. Taq polymerase의 thermostability는 PCR methodology에서 필수적인 요건이다.
PCR 증폭반응을 시작하기 위해, 그 효소를 primer가 결합된 DNA에 첨가하고 새로운 상보적인 가닥을 합성시키기 위해 반응시킨다. 그리고 나서 새롭게 합성된 가닥을 주형으로부터 분리시키기 위해 그 혼합액을 94℃로 가열시킨다. 그리고 primer들이 새롭게 합성된 가닥을 포함하여 각각의 위치에 혼성화되도록 하기 위해 냉각시킨다. 이제 Taq polymerase는 두번째 DNA합성반응을 수행하게 된다. Denaturation-hybridization-synthesis로 구성된 cycle을 반복 수행한다(대개 25-30번). 그 결과 증폭된 DNA 단편의 수많은 copy들이 합성된다. PCR실험의 마지막 단계에서, 반응 혼합액 시료를 agarose gel electrophoresis에 의해 분석한다. 단편이 증폭되어 생성된 충분한 양의 DNA를 EtBr염색 후에 band로써 확인할 수 있다. 이것은 그 자체로, 증폭된 DNA부위에 대한 유용한 정보를 제공할 수 있다. 또한 PCR산물은 정상적인 방법으로 조작된 plasmid 또는 bacteriophage vector내로 삽입되어 DNA sequencing같은 표준 기술에 의해 분석될 수도 있다.

실험관 내에서 특정 DNA를 계속 연쇄적으로 중합시켜서 엄청나게 증폭시키는 기법이 PCR이다.
Denaturation : 세포내에서는 헬리카아제가 한다. 그래서 보통은 열을 가해서 denature을 한다.
95도 이상 2분에서 5분정도 가열하면 헤어진다.
그런 다음 단일가닥으로 노출 됐으니까 프라이머를 넣어준다.
3’에서 5’ 로 읽히고 프라이머는 5’ 3’ 으로 복제가 된다.

프라이머를 붙일 때는 식혀야 한다. 그러면 프라이머가 견딜 수 있다.
또 실제로 DNA를 또 합성할 때는 좀 더 올려서 합성한다.


마지막에 내가 원하는게 나오게 된다.
실제로 내가 30사이클을 합성한다 하면 2^28승이 합성된다
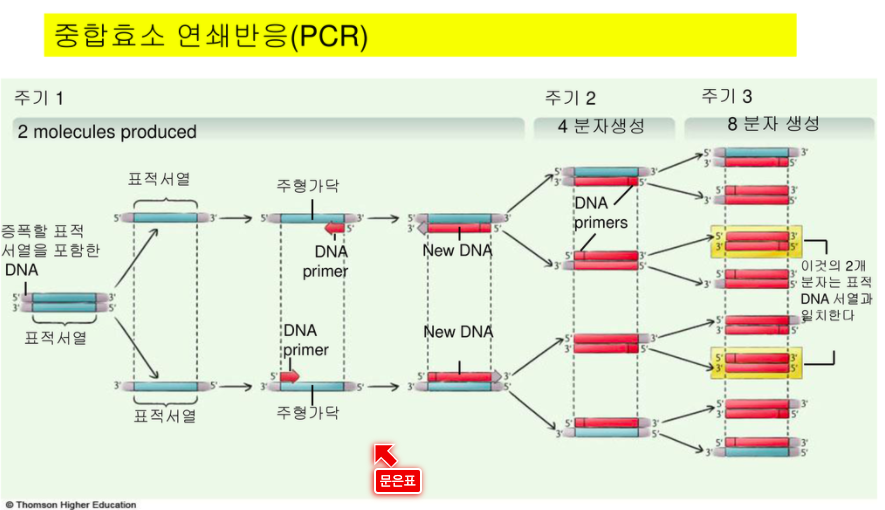
3번째 주기가 되어서야 비로소 내가 원하는 가닥이 2개 나온다.
2^(n-2)
그림 하나 더있음

그림 9.4 전기영동으로 DNA 조각 확인하기.
3번은 다른 사이즈의 DNA, 4번은 내가 원하는게 나왔지만 백그라운드 DNA도 나왔다.
PCR 조건을 확립하면 내가 원하는 정확한 DNA를 얻을 수 있다.
PCR in more detail
PCR실험을 준비하는 것이 매우 쉽긴 하지만, 그 결과가 어떤 가치를 갖기 위해선 주의깊게 계획되어야만 한다. Primer의 서열은 이 실험의 성공을 위한 결정적인 요소이다. 또한 일단 얻어진 증폭진 DNA분자를 가지고 무엇을 할 수 있느냐는 중요한 문제가 있다.
Designing the oligonucleotide primers for a PCR
Primer는 PCR실험의 성패를 좌우하는 핵심요소이다. Primer가 정확하게 design되면 그 실험은 주형분자의 target region과 대응하는 단일의 DNA단편을 증폭시킬 것이다. Primer가 부정확하게 design되면 그 실험은 실패하여 증폭이 일어나지 않거나 잘못된 단편이 증폭되거나 한 종류이상의 단편이 증폭될 것이다.
적절한 서열을 가진 primer를 design 하는 것은 문제가 되지 않는다. 그들은 주형분자의 target region의 반대쪽 서열과 맞물려야만 한다. 물론 각 primer는 주형가닥과 혼성화가 일어날 수 있도록 그것과 상보적이어야만 한다. 그리고 혼성화된 primer들의 3'말단은 서로를 향하고 있어야 한다.

피 한방울 뽑아서 DNA 추출하면 PCR 돌리면 나온다.
프라이머 서열은 헤모글로빈 유전자 서열을 우리가 알기 때문에 상보적인 걸 붙여주면 된다.
프라이머는 내가 증폭하고 싶은 부분에 있어야한다.
내가 증폭시키고 있는 부분을 포함해서 그 바깥쪽으로 디자인 해야한다.
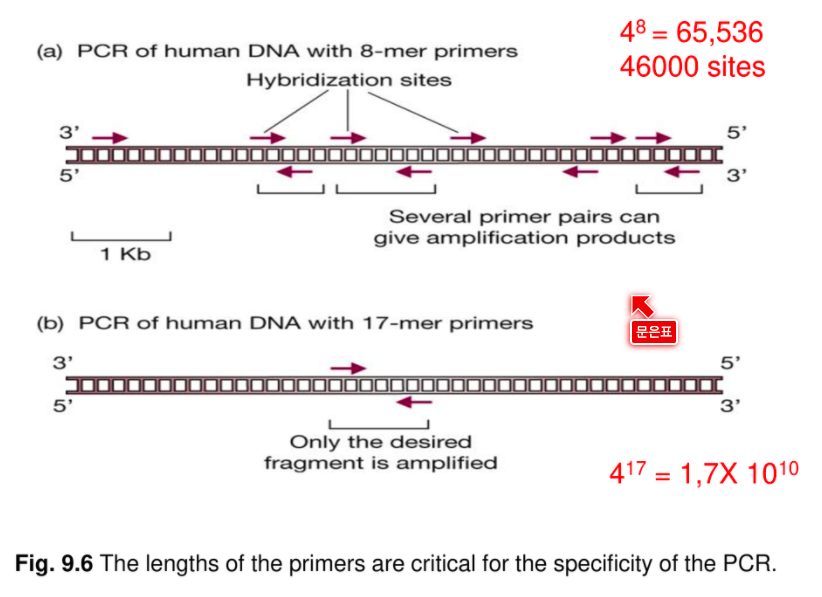
8 mer = 8개의 base pair
각각 4가지의 서열이 있으니 8 mer는 총 4^8 가지이다.
17mer짜리는 굉장히 specific해서 잘 찾아낼 수 있다.
그런데 너무 길면 PCR 합성 시간이 늘어난다.
일반적으로 17mer 정도 되면 백그라운드 없이 증폭시킬 수 있게 된다.
상세 설명
증폭될 DNA단편은 길이상 약 3kb보다 더 크지 않아야 하며 이상적인 길이는 1kb미만이다. 10kb이상의 단편들이 표준 PCR기술들에 의해 증폭될 수는 있지만 더 긴 단편일수록 증폭 효율은 더 낮고, 안정적인 결과를 얻기가 어려워진다.
Primer는 얼마나 길어야 하는가?
Primer가 너무 짧으면 그들은 non-target site와 혼성화를 이루게 되어 원하지 않는 증폭산물을 생산할 것이다. 이를 설명하기 위해, total human DNA가 8 nucleotide길이의 primer(8-mer) 한 쌍과 함께 PCR실험에 사용된다고 상상해 보자. 어떤 8 nucleotide 서열이 한번 정도 나타날 수 있는 가능한 길이는 4^8=65,536bp이다. 즉 65,536bp 개의 DNA상에 어떤 8 nucleotide와 같거나 상보적인 서열을 갖는 부위가 한 곳 정도 존재할 가능성이 있다는 것이다. Human genome은 3,000,000kb정도이며 여기에 8 nucleotide가 match할 수 있는 부위는 대략적으로 46,000곳 정도나 된다. 이것은 한 쌍의 8-mer primer는 human DNA에 대해 단일의 특이적인 증폭산물을 생산할 수 없다는 것을 의미하는 것이다.
17-mer primer가 사용되는 경우는 어떠한가? 17-mer sequence의 예상되는 빈도수는 417=17,179,869,184bp이다. 이러한 수치는 human genome의 길이보다 5배 이상 더 긴 것이다. 17-mer primer는 total human DNA에서 단 하나의 혼성화 부위를 갖는다고 예상할 수 있다. 따라서 한 쌍의 17-mer primer는 단일의 특이적인 증폭산물을 생산할 것이다. 왜 간단하게 가능한 한 긴 primer를 만들지 않는가? 그 이유는 primer의 길이는 그것이 주형 DNA와 혼성화를 이루는 속도에 영향을 주기 때문이다. 더 긴 primer일 수록 더 느린 속도로 혼성화를 이룬다. 따라서 실험동안 생산된 증폭된 분자의 수에 의해 추정되는 PCR의 효율은 primer가 너무 길 경우 감소하게 된다. 이 경우 주형분자와의 완전한 혼성화는 reaction cycle동안 허용된 시간내에 일어날 수 없다.
Working out the correct temperatures to use
PCR의 각 cycle동안에 reaction mixture는 세 가지 온도를 거치게 된다.
- Denaturation temperature(보통 94℃)
- Hybridization or annealing temperature
- Extension temperature(보통 74℃, 이는 Taq polymerase의 최적 활성 온도보다 약간 낮은 온도임)
Annealing temperature는 이것이 반응의 특이성에 또 다시 영향을 줄 수 있기 때문에 중요하다. DNA-DNA hybridization은 temperature-dependent phenomenon이다. 온도가 너무 높으면 혼성화가 일어나는 대신 primer와 주형이 분리된 상태로 남아있게 된다. 하지만 온도가 너무 낮으면 mismatched hybrids가 안정적인 조건이 되어 버린다. Mismatch가 안정적이라면 각 primer를 위한 잠재적인 혼성화 부위의 수는 엄청나게 증가될 것이고 증폭은 주형분자의 non-target sites에서 더 많이 일어날 것이다.

이 그림 기억해라.
여기에서 중요한게 Annealing이다.
짧은 프라이머를 붙이는 것.
프라이머는 유전자마다 specific 하기 때문에.

Annealing
이상적인 annealing temperature는 primer와 template사이에 혼성화가 일어날 만큼 낮아야 하지만 mismatched hybrid가 형성되는 것을 방지할 만큼은 높아야 한다. 이 온도는 primer-template hybrid의 Tm(melting temperature)을 결정함으로써 추정될 수 있다. Tm이란 정확하게 염기쌍을 이룬 hybrid가 분리될 때의 온도를 말한다. 이 온도보다 1-2℃ 낮은 온도는 정확한 primer-template hybrid가 형성되기에 충분할 만큼 낮지만 single mismatch를 가진 hybrid가 안정적으로 유지되기 위해서는 너무 높을 것이다. Tm이 실험적으로 결정될 수는 있지만 대개 간단한 공식에 의해 더 많이 계산되고 있다.

Annealing 온도 맞추기.
각각의 염기 개수를 센다.
GC 합한 갯수 x 4 + AT 합한 개수 x 2
Tm : 두가닥이 헤어지는 온도
정확하게 말하는 한분자 가지고 실험하지 않기 때문에 절반이 헤어지는 온도이다.
그래서 1도에서 2도정도 낮게 설정해준다.
Tm = (4×[G+C]) + (2×[A+T])℃
[G+C]는 primer서열에서 G와 C nucleotide의 수이고 [A+T]는 A와 T nucleotide의 수이다. 따라서 PCR실험을 위한 annealing temperature는 각 primer에 대한 Tm을 계산하여 이 수치보다 1-2℃ 낮은 온도를 이용함으로써 결정된다. 이것은 두 개의 primer가 동일한 Tm을 갖도록 고안되어야 함을 의미한다. 만일 그렇지 않으면, 한 primer에 대해서는 적절한 annealing temperature이지만 다른 primer에 대해서는 너무 높거나 너무 낮을 수도 있다.
After the PCR : Studying PCR products
PCR은 original template로서 작용하는 DNA분자에 대한 정보를 얻기 위해 다양한 방법으로 연구되는 증폭산물에 대한 일련의 실험들을 위한 출발점이기도 하다. 폭넓은 범위의 기술들이 PCR산물의 연구를 위해 고안되어 있긴 하지만, 가장 중요한 세가지 유형의 분석법들은 다음과 같다.
- Agarose gel electrophoresis
- PCR산물의 직접적인 서열분석
- PCR산물을 cloning하는 것과 관련된 분석
1. Gel electrophoresis of PCR products
PCR산물은 agarose gel에서 전기영동시킴으로써 확인될 수 있다. 증폭된 DNA를 나타내는 band는 EtBr염색을 통해 육안으로 관찰되거나 만약 수율이 낮은 경우에는 Southern hybridization에 의해 탐지될 수도 있다. 예상된 band가 없거나 추가적인 band가 존재하는 경우에는 무언가 잘못된 것이고 실험을 다시 수행해야 한다.
어떤 경우에 있어서, agarose gel electrophoresis는 PCR실험이 수행되었는지를 결정하기 위해서 뿐만 아니라 추가적인 정보를 얻기 위해 사용된다. 예를 들어 주형 DNA의 증폭된 부위에 존재하는 restriction site는 agarose gel에서 시료를 전기영동시키기 전에 PCR산물을 제한효소로 처리함으로써 결정될 수 있다. 또한 PCR산물의 정확한 크기는 주형 DNA가 증폭된 부위에 insertion이나 deletion mutation을 가지고 있는지를 결정하기 위해 이용될 수 있다.

어떤 유전자를 증폭해서 나왔다.
제한효소로 잘랐을 때

PCR은 Gene cloning을 시간을 줄여준다.

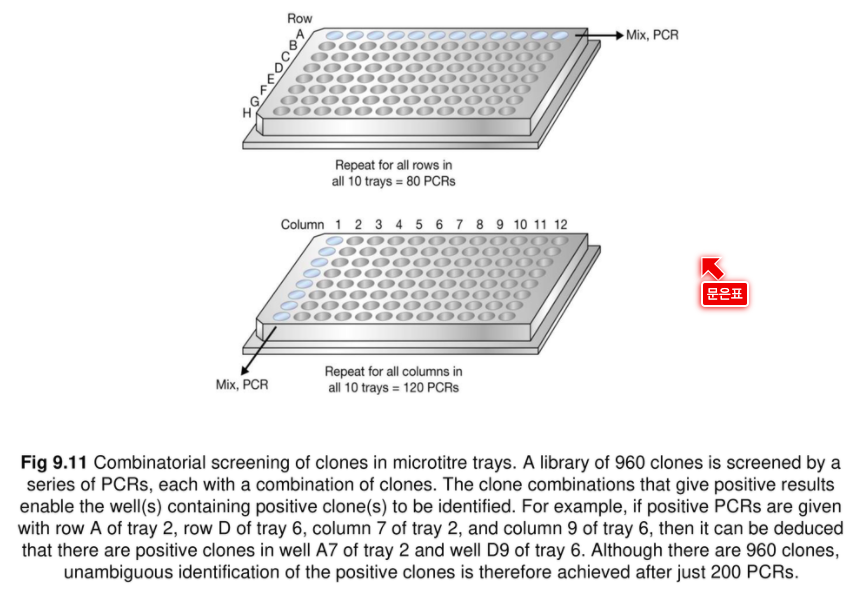
원래 첫번째 판하고 두번째 판하고 이렇게 하는데
첫번째 판과 두번째 판을 한꺼번에 묶어서 하면 더 빠르게 할 수 있다.
그럼 96개씩 묶어서 할 수 있다. 12 * 8


T가 하나 더 붙어있는 벡터를 이용한다.

BamH1으로 자르면 ExtraA도 잘려나가고
Sticky End가 생기기 때문에 클로닝이 쉬워진다.

몇 개 더 추가한다.

토퍼아이소머레이스
DNA에 결합해서 자른 다음에 떨어져나가지 않고 두가닥을 다른방향으로 꼬아서 다시 붙인다.
제한효소처럼 자르고 ligase 역할을 혼자 다한다.

A를 끝에 갖고있는 PCR의 클로닝의 예

(a) 인산기를 3’쪽에 붙인다.
(b) 3’에 이을려면 5’에 또 인산기가 있으면 안되므로 알칼라인 포스페이트를 처리해서 제거해준다.
그러면 인산에스테르 결합을 할 수 있다.
(c) s
Taq DNA polymerase는 문제점이 있다.
클로닝하기가 굉장히 어렵다는 것이다.
그걸 해결하는 방법이다 이게.

두번째 Taq DNA의 문제점은 복제실수율이 높다는 것이다.
실수가 나오면 돌연변이가 생긴다. 돌연변이가 생기면 길게 중합을 못한다.
그래서 중합된 길이가 굉장히 짧다.

Vent : 또 다른 Thermococus라는 호열성으로부터 얻은 것이고 처음부터 Recombinant를 쓴다.
Deep Vent
Pfu 다른 균주에서 얻은것
이것까진 신경 쓸 필요는 없다.

표 외우기 . 잘읽어보도록 하세요~

qPCR : PCR 한 사이클 돌고나서 얼마나 생겼는지 실시간으로 체크하는 것
증폭과 정량을 동시에 할 수 있다는 장점이 있다.


실시간 증폭하면서 양을 보고 증폭하면서 양을 보고
양을 중점적으로 본 quantitative PCR
마지막에 DNA가 관측되는 것을 본다는 점과 차이가 있다.

증폭된 DNA이다.
증폭된 DNA가 나타내는 형광
Quenching pound 형광을 억제하는 것
양쪽 끝으로 헤어지게 되면 Quencher가 형광을 억제를 못하므로
내가 원하는 DNA가 아니라면 형광이 나타나지 않는다.
내가 원하는 DNA가 증폭된다면 그만큼 비례해서 형광을 얻을 수 있다.


RT-PCR은 최초에 RNA를 이용한다.
Reverse transcriptase를 쓰고 RNA를 하나 제거해주면
PCR의 주형으로 쓸 수 있다.

각 사이클을 돌았을 때

매번 두배씩 늘어나진 않는다.

기기의 한계
Cycle수가 DNA에 반비례한걸 알 수 있다.
적은 분자일수록 더 많이 돌려야 한다.

사이클 수가 적을 때 자르면 선 아래에 있는데에선 DNA가 안 나타날 수 있다.
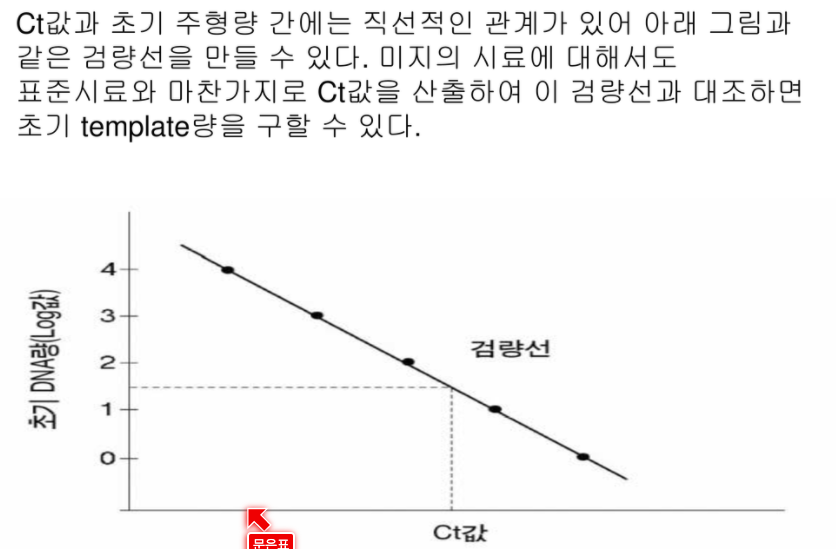
사이클이 어느정도 돌아가면서 매번 농도를 측정할 수 있게 된다.
Real time PCR은 증폭하면서 양을 잰다. 원래 30사이클을 돌려서 2^28개가 나오는 건데
'🧬 Bio > 유전공학' 카테고리의 다른 글
| 유전공학 10장 - Next Generation Sequencing (2) (0) | 2020.04.27 |
|---|---|
| 유전공학 10장 - Next Generation Sequencing (0) | 2020.04.23 |
| 유전공학 8장 (2), 9장 - 유전자 및 유전체 구조 연구 (0) | 2020.04.16 |
| 유전공학 7장 (2), 8장 - 특정 유전자의 클론을 얻는 방법 (0) | 2020.04.13 |
| 유전공학 7장 - 대장균 이외의 다른 생물체 용도의 클로닝 벡터 (0) | 2020.04.09 |