어떻게 내가 원하는 Clone을 고를 것인가?
- Direct Selection
- Gene Library = Clone Library
- cosmid에 담고 packaging하여 만든다.
- cDNA Library
- 염색체 서열은 암호화 부위만 발현된다. 발현이 안되는 유전자는 Silent gene이다.
- DNA가 아니라 mRNA를 이용하여 발현되는 유전자들만 담는 Library
- Expression Library : 단백질까지 만들어지는 Library
- cDNA cloning : mRNA는 cDNA 합성에 의해 DNA로 전환될 수 있다.
- First strand synthesis : mRNA의 poly(A) tail에 oligo(dT) primer를 결합시킨다. 역전사효소(Reverse transcriptase) 첨가
- RNA degradation : mRNA를 주형으로 상보적인 DNA가 합성되면 RNase H1를 첨가하여 부분적으로 RNA가닥을 제거한다(이 때 RNA fragments가 존재)
- Second strand synthesis : DNA pol I을 첨가하면, RNA fragment가 primer 역할을 하여 DNA 가 합성된다(ds cDNA)
- Ligation into a vector
- Transformation
- Plate out
- cDNA clone 형성
- Colony hibridization ****
- Hybrdization probing : bacterial colony나 phage plaque 내에 존재하는 재조합 DNA분자를 동장할 때 이용
- Colony를 nitrocellulose or nylon membrane으로 옮긴다
- Lysis buffer로 cell을 파괴
- Alkali + protease 처리
- 막에 DNA가 강력히 결합되도록 하기 위해 80℃에서 2시간 처리 또는 UV를 조사(UV cross linker 이용)
- Probe와 hybridization
- Washing, S1 nuclease 처리
- Apply X-ray film
- Hybrdization probing : bacterial colony나 phage plaque 내에 존재하는 재조합 DNA분자를 동장할 때 이용
- Labelling
- biotin - dUTP
- horseradish peroxidase , Luminol

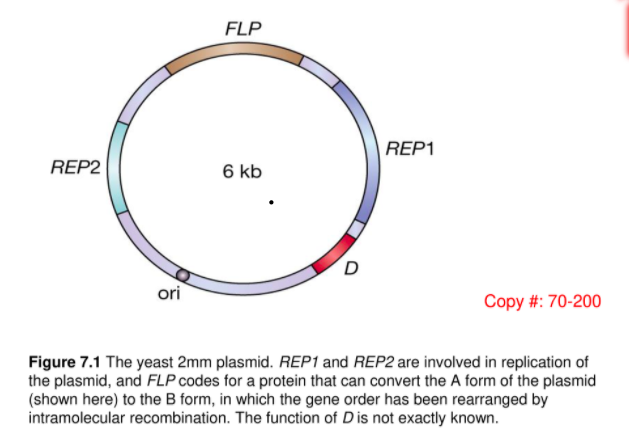
유동성 유전인자라고 불린다.
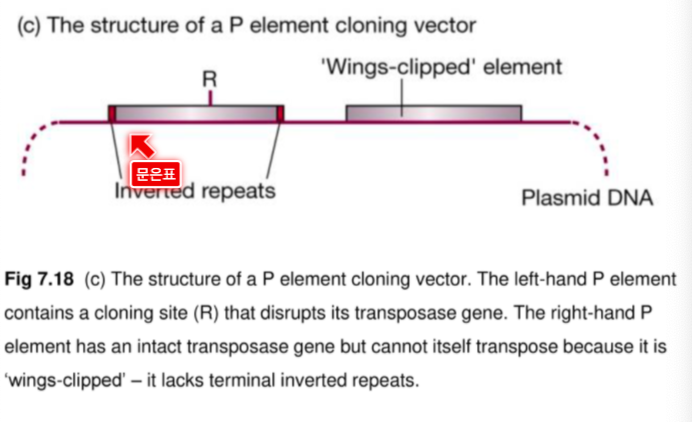
이 트랜스포존은 양 끝에 inverted repeat 가 있다. (트랜스포존은 P요소의 P특성을 담당하며 야생 파리에서만 발견된다.)
역 반복 서열
Transpoase : 자기 양끝의 반복서열을 자른 다음에 새로운 곳으로 이동시킨다.
P element를 가지고있는 플라스미드를 도입하면 P element가 염색체로 이동한다.
P element에 우리유전자를 삽입하면 초파리의 염색체로 이동시킬 수 있다.
만약 이 플라스미드에 삽입을 해서
자기자신은 역 반복서열이 없기 때문에 Transposase가 망가진 것만 집어 넣는다.
R 쪽 부분만 들어있는 형질전환체를 얻을 수 있다.

결국은 표적서열이 양 말단에 생성이 된다.

표적을 잘라
자기 말단에 있는 역반복서열 바깥에
Transposom 몰라도 상관없는것
7.3 Cloning Vectors for Animal Cells
Direct gene transfer가 가능하지만 그 효과는 매우 낮다. YAC과 동일한 개념으로, 포유동물의 인공염색체도 또한 가능하긴 하지만 실제로 보고된 바는 아직 없다. 따라서 대부분의 조작실험은 virus에 기초한 vector에 의존하고 있다.
7.3.1 Vectors based on animal viruses

레트로 바이러스 많이 사용한다.
뒤에 실제 응용에서 자세하게 한다.
많이 사용하는 이유는 끼어들어가는 자리가 다양해서 다양한 것을 얻을 수 있고
오랫동안 유지되기 때문이다.

박테리아의 플라스미드를 그냥 쓰거나 유전자의 조각을 대량으로 때려넣거나 한다.
우연히 생식세포에 된건 계속 유전이 된다.
생식세포에 들어간 non-chimera를 추려낼 수 있다.

동물에다가 집어 넣으면 멀티플 카피로 줄줄이 된다.
다량의 유전자 산물을 만들어낼 때 유리하다.

배아 줄기세포 만드는거
여기까지가 7장의 내용이다.
7장은 유전공학의 핵심기법 gene 클로닝
재조합체를 만들고
숙주에 집어넣고
제대로 들어간 형질전환체를 다량으로 증식시키는 것 = Gene cloning
운반체를 다양한 숙주별로 어떻게 개량되어왔는가를 자세하게 배웠다.
8장
How to Obtain a Clone of a Specific Gene
8.1 The Problem of Selection
가장 간단한 생물체인 E. coli 조차도 수천개의 유전자를 가지고 있으며, 따라서 전체 세포의 DNA가 제한 효소로 처리되면 원하는 유전자를 가진 단편뿐만 아니라 다른 유전자를 가진 또 다른 많은 단편들이 생기게 될 것이다. Ligation 반응동안 개개의 단편을 선별할 수 없으며 서로 다른 DNA조각을 가진 수많은 재조합 DNA분자들이 생성될 것이다. 그 결과 형질전환과 plating한 후에 다양한 재조합 clone을 얻게 될 것이다.
① Restriction of a large DNA molecule
② The resulting recombinant DNA molecules
③ All will produce colonies
8.1.1 There are two basic strategies for obtaining the clone you want


각각 다른 부위의 유전자를 가지고 있는 컬렉션이 얻어진다.
이것들을 플레이트 위에 기르게 되면 온갖가지 조각을 갖고 있는 것을 얻게된다.
이게 Genome library가 될 수 있다.
그래서 모든 형질전환체가 내가 원하는 형질전환체가 아니다.
그래서 이 장에서는 배우는 것은 어떻게 내가 원하는 형질전환체를 고르느냐이다.
상세 설명
원하는 clone을 얻을 수 있는 많은 방법들이 있지만, 모두 두 개의 범주로 나눌 수 있다.
1) Direct selection for desired gene
원하는 유전자를 가진 clone만 colony를 형성하도록 하는 방법
모든 선별은 plating 단계에서 수행됨.
2) Identification of the clone from a gene library
우선 shotgun cloning 실험을 통해 clone library를 만들고 난 후에 그 중에서 원하는 clone을 찾는 방법
* clone library : 특정 생물체에 존재하는 유전자들을 모두 갖고 있는 clone들의 집합체
Direct selection은 신속하고 비교적 명확하기 때문에 선호되는 방법이다. 그러나 모든 유전자에 응용할 수는 없다. 그러므로 clone identification 기술이 매우 중요하며 특히 많은 생물체의 완전한 genomic library가 현재 유용하기 때문에 더욱 그렇다.
8.2. Direct Selection

첫번째 방법은 Direct selection 이다.
적절한 배지만 만들 수 있으면 내가 원하는 것을 얻을 수 있다.
하지만 이런 경우는 흔치않다.
조작된 유전자를 선별하기 위해선 원하는 recombinant만 자랄 수 있는 agar medium에 transformant를 plating해야 한다. 그러면 생성된 colony만 원하는 재조합 DNA분자를 가진 세포들로 구성되어 있을 것이다.
<항생제 저항성 유전자의 cloning -ex,plasmid R6-5>
다 깔아놓고 골라야 한다 사실.


8.2.1 Marker rescue extends the scope of direct selection
Direct selection이 단지 항생제 저항성 유전자를 cloning하기 위해서만 이용된다면, 매우 제한적인 실험만 하게 될 것이다.
다행히 그 기술은 형질전환을 위한 숙주로써 E. coli 의 돌연변이 균주를 사용함으로써 실험의 폭을 넓힐 수 있다.
케나마이신 내성유전자가 있다면
제한효소로 다 자른다.
그러면 얻어졌을 때 배지에다가 케나마이신을 넣어주면 내성유전자 갖고 있는 것만 산다.
이런게 많지 않다.

이스트 지놈이 나와서 (a) 에 유전자를 가지고있다.
그러면 이스트에 라이브러리를 만든다.
trpA 를 가지고 있는 거만 산다.
(d) 찾기 피피티
8.2.2 The scope and limitations of marker rescue
Marker rescue가 많은 유전자의 clone을 얻기 위해 이용될 수 있지만, 그 기술에는 두 가지 limitation이 있다.
1) 돌연변이 균주는 그 유전자를 위해 유용해야만 한다.
2) 야생형만 생존할 수 있는 배지가 필요하다.
일반적으로 marker rescue는 생합성 효소를 암호화하고 있는 유전자들을 위해 응용될 수 있다. 왜냐하면 이들은 최소배지에서 선별될 수 있기 때문이다.
8.3 Identification of a Clone from a Gene Library

8.3.1 Gene libraries
Genomic library란 특정 생물체내에 존재하는 모든 단일 유전자를 포함할 만큼 충분한 수의 clone의 집합체를 말한다. Genomic library는 먼저 total cell DNA를 분리한 후, partial digestion시켜 생성된 단편들을 λ replacement vector, cosmid, YAC 같은 적합한 vector내부로 cloning함으로써 만들 수 있다.


* Clone library 만드는 법
Total cell DNA
↓ partial digestion
DNA fragments
↓
Ligate into cosmid
↓
in vitro packaging
↓
infect E. coli
↓
Plate out
↓
Clone library = genomic library = gene library
죽이는 배지를 얻을 수 없으면
모든 애들을 살려놓고 깔아놓고
뒤져가야 된다.
그렇기 때문에 진모을 작은 조각으로 자르면 너무 많은 클론을 뒤져봐야한다.
그래서 큰 용량의 벡터가 필요하게 된다. 35kb
라이브러리는 코스미드에다가 만들 수 있다.
기본적으로 플라스미드 벡터인데
패키징 믹스를 넣어주면 50kb되면 람다로 인정이 돼서
플라스미드 replication origin으로 인식돼서 collection이 만들어지면 gene library가 만들어진다.
몇개를 깔아야 되는지는 계산해서 깔아야 한다.
8.3.2 Not all genes are expressed at the same time
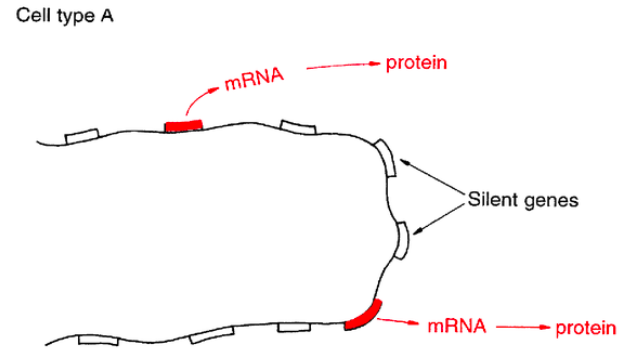
대부분의 다세포 생물체의 특징은 개별세포의 분화이다. 예를 들면 인간은 수많은 다양한 세포 유형으로 구성되어 있다(뇌세포, 혈액세포, 간세포 등). 물론 각 세포는 동일한 유전자 모두를 가지고 있지만 다양한 세포 유형에 따라 각기 다른 유전자들이 발현되며 나머지 유전자들은 발현되지 않는다.
비교적 소수의 유전자들만이 어떤 유형의 세포에서 발현된다는 사실은 조작될 물질이 DNA가 아니라 mRNA인 경우에 library 제조에 이용될 수 있다. 이렇게 발현되는 유전자들만이 mRNA로 전사되며 mRNA가 출발물질로서 사용될 경우, clone들은 세포내 전체 유전자들 중 선택된 것만을 가지게 될 것이다. mRNA를 이용하는 cloning방법은 원하는 유전자가 개별 세포유형에서 높은 비율로 발현되는 경우에 특히 유용하다.


인간의 경우 2%만 유전자 부위이다.
나머지는 유전자 정보와 관련이 없다, 코딩 부위가 아니다. 암호화부위
그래서 우리가 굳이 다 잘라서 다 뒤져볼 필요는 없다.
그래서 다른 라이브러리를 생각해 본다.
그게 뭐냐면 두번째 라이브러리인 expression library이다.
유전자가 발현이 되면 mRNA를 거쳐서 단백질이 된다.
발현되는 유전자는 따로 있다. 발현 안되는 유전자는 Silent gene 이다.
발현되느냐 안되느냐는 mRNA로 알 수 있다.
8.3.3 mRNA can be cloned as complementary DNA
★★★★★★★
mRNA는 그 자체로 cloning vector 내부로 결합될 수 없다.
하지만 mRNA는 cDNA합성에 의해 DNA로 전환될 수 있다.
이 방법의 핵심은 RNA가닥과 상보적인 DNA가닥을 합성하는 역전사효소(Reverse transcriptase)이다.
일단 cDNA가 합성되면 이 혼성화된 분자를 RNase H1로 partial digestion시킨다.
남아있는 RNA단편들은 제2의 cDNA가닥을 합성하는 DNA polymerase I을 위한 primer역할을 하여, 그 결과 vector 내부로 연결되어 cloning될 수 있는 이중가닥의 DNA 단편이 만들어 진다.


mRNA를 다 분리한다.
이 핵산을 분리를 하면 tRNA 등 다 있는데
mRNA만 분리한다.
mRNA에만 있는 폴리테일을 분리를 해서
reverse transcriptase를 이용하면 (RNA를 주형으로하는 DNApolymerase)
..
그래서 RNA를 제거한다.
RNase H1
완벽하게 지우는건 아니라 RNA를 약간씩 남긴다.
왜?
그곳을 프라이머로 해서 두번째 가닥을 합성하기 위해서
그래야 더블스트랜드 DNA가 된다.
오카자키 조각에서 착안 한 것이다.
그다음 double strand가 만들어지면 여기에다가 sticky end 링커를 붙인다.
이렇게 만든 DNA는 cDNA이다.
DNA complimentary to RNA
First strand를 주형으로해서 second strand를 만들면 cDNA
이것을 적절한 벡터에 붙인다
간세포에서 발현된 모든 mRNA를 모두 발현하게 된다.
(e) 미완성 씨앗에서 분리된 모든 mRNA로 만든 cDNA
4개만 Gliadin clone 이다.
토탈 mRNA 중 Gliadin이 35%이다.
내가 찾고싶은 유전자가 어떤 조직에서 굉장히 많이 발견된다
그러면 그 조직의 mRNA를 분리해서 cDNA 라이브러리를 만드는 것 유용하다.
* cDNA cloning
① First strand synthesis : mRNA의 poly(A) tail에 oligo(dT) primer를 결합시킨다. 역전사효소(Reverse transcriptase) 첨가
② RNA degradation : mRNA를 주형으로 상보적인 DNA가 합성되면 RNase H1를 첨가하여 부분적으로 RNA가닥을 제거한다(이 때 RNA fragments가 존재)
③ Second strand synthesis : DNA pol I을 첨가하면, RNA fragment가 primer 역할을 하여 DNA 가 합성된다(ds cDNA)
④ Ligation into a vector
⑤ Transformation
⑥ Plate out
⑦ cDNA clone 형성
Genomic library -> cDNA library -> Expression library
cDNA library : 발현된 mRNA를 한 콜렉션
Expression library : 유전자가 발현됐다는 것은 단백질은 만드는 것이기 때문에 단백질까지 만들어지는 라이브러리
4. Methods for clone identification
일단 적절한 library가 준비된 후에는, 수많은 방법들이 원하는 clone을 동정하기 위해 적용될 수 있다. 이들 방법들 중 일부는 조작된 유전자의 번역 산물을 탐지하는 것에 기초하고 있지만 대개는 정확한 재조합 DNA분자를 직접 동정하는 것이 더 쉽다. 이것은 hybridization probing이라는 기술에 의해 수행된다.
4.1 Complementary nucleic acid strands hybridize to each other
어떤 두 개의 단일가닥의 핵산 분자들은 서로 염기쌍을 형성할 수 있다. 그 결과 혼성화된 분자는 가닥 간 결합의 수가 적기 때문에 불안정하다. 그러나 그 가닥들이 상보적이라면 추가적인 염기쌍이 형성되어 안정적인 이중가닥 분자를 형성할 것이다. 이것은 단일가닥의 DNA분자 사이에서 일어날 뿐만 아니라 단일가닥의 RNA분자들 사이, 그리고 단일가닥의 DNA와 RNA분자 사이에서도 일어난다.
핵산 혼성화는 DNA 혹은 RNA probe가 원하는 유전자와 상보적인 경우에 특정 recombinant clone을 동정하기 위해 이용될 수 있다.

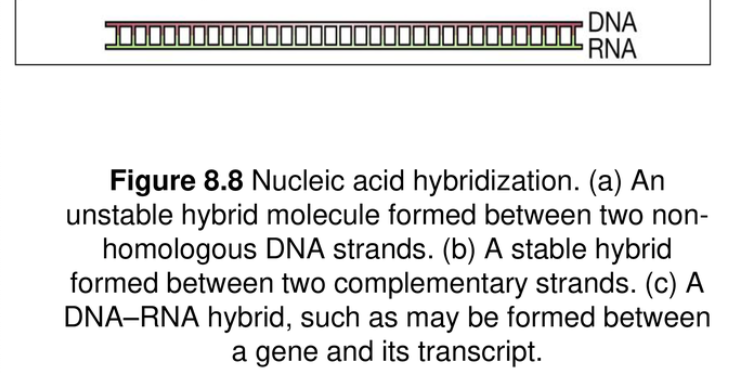
(c) 에는 수소결합에 의해 붙어있다.
강하지 않기 때문에 열을 가하면 분리된다.
Hybridaization
(a) 상보성이 없기 때문에 잘 떨어진다.
(b) 온도를 좀더 높여줘도 붙어있다.
내가 가지고 있는 것이 DNA조각이다. 이걸 가지고 유전자를 완벽하게 가지고 있는 클론을 찾는다던지
내가 가지고 있는 조각과 상동성이 있는 유전자를 찾는다던지 할 때
Hybridization을 쓴다.
대신 이걸 쓰려면 DNA 조각이 있어야한다
Probe hybridization
부합화(hybridization)란 두 개의 단일가닥 핵산이 상보적인 결합에 기초한 상호작용을 통해 이중가닥(duplex)을 형성하는 것으로, 분리되어 있는 상보적인 가닥이 가역적이면서, 염기순서특이적인 결합이 이루어지는 과정을 의미한다. 일반적으로 표지자가 부착되어 있는 한쪽 가닥을 탐색자(probe), 부합화로 형성된 이중가닥을 hybrid라고 하며, DNA-DNA, RNA-RNA, DNA-RNA 형태의 이중가닥 구조의 hybrid가 가능하다. 검출하고자 하는 핵산과 탐색자간 부합반응을 최적화하거나, 검출 성능을 향상시키고 해석을 용이하게 하기 위해 다양한 형식의 부합화 기법이 개발되어 왔다. 하지만 부합화 기법마다 장단점이 있으며, 매 검사마다 양성, 음성 대조 물질을 사용하여 부합반응의 적절성, 비특이적 반응 여부 등을 확인하는 것이 필요하다.
4.2 Colony and plaque hybridization probing
★★★★★★★
Hybridization probing은 bacterial colony 또는 bacteriophage plaque 내에 존재하는 재조합 DNA분자를 동정하기 위해 이용될 수 있다.

a) Colony 또는 plaque를 nitrocellulose 나 nylon membrane으로 이동시킨다.
b) DNA만을 남기고 다른 모든 물질을 제거시키기 위한 처리를 한다. 보통, 이 처리중에 DNA분자가 변성되어 이중가닥에서 각 가닥 사이의 수소결합이 끊어진다.
b) 이 단일가닥 분자를 membrane에 강력하게 부착시킨다.
- nitrocellulose membrane인 경우, 80℃에서 두시간 처리
- nylon membrane인 경우, 자외선 조사
DNA 분자의 sugar-phosphate backbone이 막에 강력하게 결합되고 염기는 free한 상태가 된다.

c) Nick translation 이나 end-filling 또는 random priming으로 표지된 probe를 열로써 변성시킨 후에 핵산 혼성화를 촉진시키는 화학 용액에서 막과 반응시킨다.
5) 혼성화가 일어나도록 하기 위한 시간이 경과한 후에 결합되지 않은 probe를 제거시키기 위해 S1 nuclease를 처리하고 washing을 한다.
6) Autoradiography에 의해 혼성화가 일어난 위치를 확인한다.
내 DNA조각이 있다면 별을 단다. Labeling을 한다.
DNA에 붙여보면 내가 가지고있는 유전자 조각과 상동성을 갖고있는 클론에 붙는다.
대충 붙는거 같지만 온도를 높여가면서 워싱을 하면 우연히 들러붙은 애들을 다 씻기고
충분히 동일한 유전자를 같는 애들은 완전하게 붙어있다.
그 후 X-ray 필름을 얹으면
잘 붙어있는 클론 위치에만 감각이 된다.
탐침 = probe
* Colony hybridization
① Colony를 nitrocellulose or nylon membrane으로 옮긴다
② Lysis buffer로 cell을 파괴
③ Alkali + protease 처리
④ 막에 DNA가 강력히 결합되도록 하기 위해 80℃에서 2시간 처리 또는 UV를 조사(UV cross linker 이용)
⑤ Probe와 hybridization
⑥ Washing, S1 nuclease 처리
⑦ Apply X-ray film
그러나 방사능 표지 방법은 연구자에게 해를 끼칠 수 있고, 방사성 폐기물의 처리와 관련된 문제를 가지고 있다. 따라서 hybridization probe를 non-radioactive manner로 표지시키기도 한다.
1) Labelling with a biotinylated nucleotide
Avidin이라 불리우는 단백질과 높은 친화도를 가진 유기분자인 biotin과 함께 반응시킴으로써 변형시킨 dUTP를 사용하는 방법.
혼성화 후에 biotinylated probe와 결합된 위치는 fluorescent marker인 avidin과 반응시킴으로써 탐지될 수 있다. 이 방법은 radioactive probing만큼이나 민감하여 그 사용이 증가하고 있다.
2) Labelling with horseradish peroxidase
Probe DNA가 horseradish peroxidase와 복합체를 형성하도록 한다. 결과는 그 효소가 luminol을 분해하여 chemiluminescence를 발산시키는 능력을 통해 탐지되며, 이 signal은 autoradiography와 유사한 방법으로 일반 사진 film상에 기록될 수 있다.

probe를 어떻게 Labling 하느냐
구멍을 만든다음에 DNA pol을 넣어주고 P-dATP(동위원소 뉴클레오타이드)를 넣어주면 된다

짧은 핵소머, 염기6개짜리가 여기저기 가서 붙는다
얘는 프라이머로 쓸 수 있고
동위원소를 넣어주면
프라이머로부터 중합이 시작되면서 동위원소로 표지가 된 것이 만들어진다.
동위원소는 맨눈으로 보면 안되고 X-ray film을 얹어주면 까맣게 감광된 필름이 얻어진다.
내 프로브DNA를 가지고있는 클론이 발견된다.
'🧬 Bio > 유전공학' 카테고리의 다른 글
| 유전공학 9장 - 중합효소 연쇄 반응(PCR:The polymerase chain reaction) (0) | 2020.04.20 |
|---|---|
| 유전공학 8장 (2), 9장 - 유전자 및 유전체 구조 연구 (0) | 2020.04.16 |
| 유전공학 7장 - 대장균 이외의 다른 생물체 용도의 클로닝 벡터 (0) | 2020.04.09 |
| 유전공학 6장 - 대장균(E. coli) 용도의 클로닝 벡터 (0) | 2020.04.06 |
| 유전공학 5장 - Introduction of DNA into living cells (0) | 2020.04.02 |