1.1. Yeast에 형질전환체 넣기
- YEp13 : Yeast Episomal plasmid
- shuttle vector : 2가지의 replication origin 같기 때문에
- Ecoli 에서 증식할 수도 있고, Yeast 에서도 증식할 수 있다.
- selection marker
- ampR
- tetR
- LEU2 gene : LEU2가 있어야 배지에서 살아남을 수 있다.
- LEU2가 없는 mutant를 host cell로 해서 선별한다.
- 호스트는 2가지이다 : Ecoli, Yeast ****
- Homologous recombination : YEp13가 LEU2 유전자로 인해 Yeast chromosome으로 recombination이 된다.
- YIp5
- pBR322를 가지고 있고 대장균에서 증식 가능
- Selection maker : URA3 gene
- 반드시 삽입 되어야만 Yeast 내에 존재할 수 있다.
- YRp7
- Selection marker : TRP1 gene
- TRP1 gene 옆에 Yeast cromosome origin이 있다.
- Replication origin이 있어서 독립적인 plasmid로 복제 가능
- 형질전환 효율 (Transformation frequency)
- YEp > YRp > YIp
- Copy number
- YEp : 20 - 50
- YRp : 5 - 200
- YIp : 1
- 안정성 : YIp
- YAC (Yeast Artificial Chromosome) ****
- SUP4 gene : SnaB1 제한효소 / SUP4(+) = red (non-recombinant) / SUP4(-) white (recombinant)
- URA3 gene
- TRP1 gene : 옆에 Replication origin 있다.
- CEN4 gene : 염색체 분리
- TEL gene : BamH1 제한효소 / 텔로미어 서열
1.2. 식물세포에 형질전환체 넣기
- Agrobacterium tumefaciens
- Agrobacterium 세균(뿌리혹박테리아)이 식물염색체에 recombinant 된다.
- 상처부위에 감염되면 세포분열 된다 : 식물의 암
- Ti plasmid : 감염 후 plant chromosomal DNA내부로 통합된다.
- T-DNA (Transfer DNA)
- 식물염색체로 이동하는 부위
- 식물세포내에서 발현되는 유전자를 가지고 있다(사이토카닌, 옥신).
- 형질전환된 세포의 암적인 특징 담당
- Virulence region : T-DNA transfer에 관여
- Host specificity region
- T-DNA (Transfer DNA)
- 특징
- Size : 15 - 30kb
- Host specific (숙주특이성)
- 식물성장호르몬 옥신, 시토카인 가지고 있다.
- 과정
- Plant cell의 물리적인 상처
- phenolic compound 분비
- Agrovacterium tumefaciens가 phenolic compound 인식
- T-DNA 이동
- 숙주 chromosome에 통합
- Opine 합성 (자신의 목적을 위해 식물세포를 유전학적으로 조작)
- pBIN19 : T-DNA 양 끝을 인식하는 반복서열(25bp)을 이용하여 만든 벡터
- 식물 바이러스
- RNA genome 가지기 때문에 유용하지 않다.
- CaMV : Size limitation, Narrow hos range
- Geminivirus : Rearrangement가 많아서 변이가 쉽다.
cosmid 벡터 = cos site + λ
사이즈도 굉장히 작아졌고 재조합 했을때 ligation 줄줄이 붙일 수 있다.
cos site로 잘라서 재조합 람다를 만들 수 있다.
거의 45~50kb의 큰 DNA를 삽입할 수 있다.
람다로 패키징 해서 숙주에 섞으면 람다가 감염을 잘하기 때문에 효율적으로 잘 집어 넣는다.
숙주에 넣으면 플라스미드처럼 증식을 한다.
대용량 벡터의 시발점인 코스미드 벡터를 가지고 Genomic library를 만들 수있게 되었다.
어떤사이즈의 지놈을 가진 개체의 라이브러리가 있을 때, 내가 원하는 유전자를 가진 클론을 찾으려면 몇개를 뒤져야 하냐?
공식을 외울 필요는 없는데 어떤 것에 의해서 달라질 수 있는지
몇 퍼센트의 확률로 계산할까. 내가 얻고자 하는 DNA의 genome 사이즈,
어떤 크기로 잘랐느냐 뒤져봐야할 클론이 줄어들게 된다.
대부분의 cloning 실험은 숙주로서 E. coli 와 함께 수행되며, 매우 다양한 cloning vector 들이 이 생물체에 대해 유용하다. Cloning 실험의 목적이 유전자 구조와 기능 같은 분자생물학적인 기본적 특징을 연구할 때 특히 그렇다. 그러나 어떤 경우에는 gene cloning 실험을 위해 다른 숙주를 이용하는 것이 바람직할 수도 있다. 이것은 그 목적이 유전자 연구가 아니라 중요한 대사산물(ex, hormone)의 합성을 조절하거나 향상시키고, 또는 생물체의 특징을 변화시키는(ex, 제초제 저항성 농작물에 도입시키는 등) cloning 기법을 사용하는 생물공학분야에서 특히 그렇다. 따라서 E. coli 이외의 다른 생물체를 위한 cloning vector를 생각해야 한다.
2. 7.1 Vectors for Yeast and Other Fungi
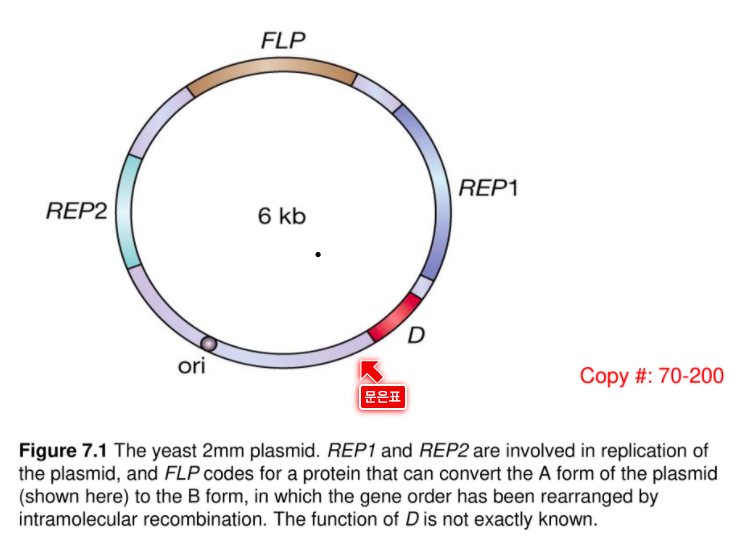
Yeast Saccharomyces cerevisiae 는 생물공학에서 가장 중요한 생물체 중 하나이다. Yeast는 양조와 제빵에서 중요한 역할을 할 뿐만 아니라, 조작된 유전자로부터 중요한 의약품을 생산하기 위한 숙주생물체로서 이용되고 있다. Yeast를 위한 cloning vector 들의 개발은 S. cerevisiae의 대부분 균주에 존재하는 plasmid인 2㎛ circle의 발견에 의해 자극되었다.
2.1. 7.1.1 Selectable markers for the 2㎛ plasmid
2㎛ circle은 크기가 6kb이고 yeast cell내에서 copy number는 70~200 사이이다. 복제는 plasmid origin을 이용하고, 복제를 위해 필요한 효소는 plasmid의 REP1, REP2 에 암호화되어 있는 단백질, 그리고 일부는 숙주세포로부터 공급받는다. 따라서 2㎛ circle은 cloning vector로서 이상적이다.
그러나 2㎛ circle 이 cloning vector로서 완벽한 것은 아니다. 우선 selectable marker가 없다. 최근의 yeast cloning vector들은 methotrexate와 copper 같은 저해제에 대해 저항성을 부여하는 유전자를 가지고 있는 것이 있긴 하지만, 대부분은 아미노산 생합성과 관련된 효소를 암호화하고 있는 yeast gene이 이용된다. 일례로 pyruvate를 leucine으로 변환시키는 효소들 중 하나인 β-isopropyl malate dehydrogenase를 암호화하고 있는 LEU2 유전자를 이용한다.
Selectable marker로써 LEU2 를 이용하기 위해선, 숙주생물이 non-functional LEU2 유전자를 가진 돌연변이체 이어야만 한다. 그러한 leu2- yeast는 leucine을 합성할 수 없고 따라서 성장배지에 leucine이 공급되는 경우에만 생존할 수 있다. Cloning 실험에서, 세포는 최소배지(추가된 아미노산이 없는)에 plating 된다. 형질전환된 세포만이 생존할 수 있고 colony를 형성할 수 있다.
원칙적으로 세균에만 들어있다.
진핵세포는 플라스미드가 없다.

자연계에서 replication origin이 있다. 독자적으로 복제할 수 있다.
이 플라스미드는 진핵세포가 가지고 있기 때문에 이걸 이용한다.

모든 플라스미드 벡터에서 중요한것 : 선별 마커이다.
엠피실린 테트라사이드는 원핵세포에만 영향을 주기 때문에 진핵세포인 Yeast에는 활용할 수 없다.
엠피실린이나 테트라사이클린이 배지에 있어도 얘내들은 영향을 받지 않는다.
생합성 과정이 망가진 이스트를 숙주로 쓴다.
(a)
LEU2- 자기 스스로 류신이라는 것을 만들 수 없다.
배지에다가 류신을 빼면 얘는 죽게된다. 류신이 있을 때만 살게 된다.
얘를 살려서 콜로니를 얻을려면 배지에 반드시 류신을 넣어줘야한다.
류신을 빼면 호스트 자체는 죽게된다.
(b) 온전한 LEU2 유전자를 넣어준다. 형질전환체
살아남는 애는 백만개 중에 살아남아도 얘를 가진 애만 살아남는다.
Selection power가 있어서 ㅎ 선별 잘 할 수 있다.
2.2. 7.1.2 Vectors based on the 2㎛ circle -yeast episomal plasmids

상세 설명
****************************************************************************************************************************************
2㎛ circle로부터 유도된 vector를 yeast episomal plasmid 또는 YEp 이라 부른다. 일부 YEp은 완전한 2㎛ circle을 가지고 있고, 나머지는 단지 2㎛ 의 replication origin 만을 포함하고 있다.
- shuttle vector
- 2㎛ 의 replication origin
- selectable LEU2 gene ★★★★★★★
- entire pBR322 sequence (ampR, tetR)
⇒ 따라서 yeast 와 E. coli 모두에서 복제할 수 있고 선별될 수 있다.
host cell로서 leu2- mutant를 이용
leu2 2㎛ circle을 leu2- mutant에 형질전환 시킴
-grow at no leucine medium → transformant
-can not grow no leucine medium → non-transformant
****************************************************************************************************************************************
첫번째가 Yep13
얘는 박테리아의 플라스미드로 구분하려고 쓰는 거다.
원칙적으로 뒤에 써준다 p
pBR322라는 박테리아의 플라스미드에
ampR tetR 이부분은 pBR322 대장균 부분이다.
표시는 안돼있지만 이 둘 사이에 replication origin이 있다.
대장균 벡터 플라스미드 이다. 세 개를 가지고 있다. → ampR, tetR, replication origin
2 마이크로미터 DNA는 이스트의 Replicaion origin이 있다.
복제될 때 얘를 이용한다
Yeast의 유전자는 LEU2 유전자를 갖는다. ★★★★★★★
pBR의 유전자는 E.coli에 넣어도 증식될 수 있다.
YEp13
- 얘는 셔틀 벡터이다. 대장균에서 증식시키다가 Yeast에다가도 쓸 수 있다는 것이다.
두가지를 다 이용할 수 있을때 셔틀벡터이다. - 왜 셔틀벡터냐: 두가지의 replication origin을 가지고 있기 때문이다.
- 플라스미드 벡터는 Selection marker가 있어야 된다고 하는데 두 개의 항생제 내성 유전자가 있고 Yeast에서도 작동하는 Selection Marker인 LEU2유전자이다.
상세 설명
****************************************************************************************************************************************
Shuttle vector를 사용하는 이유는 형질전환된 yeast colony로부터 재조합 DNA분자를 회수하기 어려울 수도 있기 때문이다. 이러한 문제는 yeast vector가 yeast chromosome 내부로 통합되는 경우 발생하며, 이 경우 재조합 DNA분자의 순수분리는 불가능하다. 따라서 yeast에서 cloning 실험을 할 때, 표준적인 실험방법은 처음에 cloning 실험을 E. coli 와 함께 수행하여 recombinant를 선별하고 난 후, recombinant plasmid를 분리하여 특징을 결정하고, yeast 내부로 그 correct molecule을 도입시키는 것이다.
****************************************************************************************************************************************
그래서 대장균에도 넣어도 되고 Yeast에 넣어도 된다.

우리가 원하는 유전자 조각이 아닌 다른 조각이 들어갈 수 있고 벡터가 닫힌 것들이 들어갈 수 있다.
그러니까 recombinant DNA를 고르는게 어렵다.
따라서 재조합 DNA을 찾는 것은 대장균에서 먼저 한다.
E.coli 클론을 얻으면 대량으로 증식해서 그때 Yeast에 때려 넣는다.
그러면 훨씬 쉽게 형질전환된 Yeast 클론을 얻을 수 있다.
그래서 셔틀벡터가 필요하고
호스트는 두가지이다. E.coli, Yeast ****
2.3. 7.1.3 A YEp may insert into yeast chromosomal DNA
상세 설명
****************************************************************************************************************************************
'Episomal' 이라는 말은 YEp 이 독립적인 plasmid 로서 복제할 수 있다는 것을 가르킬 뿐만 아니라, yeast chromosome 의 한 곳으로 통합될 수 있다는 것을 암시하는 것이다. 통합은 selectable marker 로서 vector에 존재하는 유전자가 yeast chromosome에 존재하는 돌연변이 유전자와 매우 가까운 상동성인 경우 일어난다. 예를 들어 YEp13에서, 재조합은 plasmid LEU2 유전자와 yeast mutant LEU2 사이에서 일어날 수 있다. 그 결과 전체 plasmid가 yeast chromosome의 한 부위 내부로 삽입된다. 그 plasmid는 통합된 상태로 유지될 수도 있고 또는 나중에 재조합이 일어나서 다시 excision되기도 한다.
****************************************************************************************************************************************

이스트 숙주에 들어가도 있다.
mutated LEU2와 LEU2는 상동성이 있어서 상동체 조합이 일어난다
Homologous recombination
**************************************************************************************************************************
2.4. 상동 재조합(Homologous Recombination)
전통적으로, 상동 재조합(homologous recombination)은 유전자 제거를 일으키는 중요한 방법이었다. 이 방법은 원하는 돌연변이 결과물을 포함하는 DNA 구조물을 먼저 만들어야 한다. 녹아웃을 목적으로 하는 경우, 전형적인 경우에 녹아웃 유전자의 위치에 약제내성 마커가 포함된다. 이 구조는 또한 목표 유전자배열에 최소 2kb크기의 상동(homology)을 포함한다. 이 구조물은 미세주입(microinjection)이나 전기천공(electroporation)을 통해서 (줄기) 세포에 전달된다. 이 방법은 그 이후 세포 자신의 수리 메커니즘에 의존하는데, 이는 기존의 DNA에 DNA 구조물을 재조합하는 것이다. 이 결과로 유전자 배열이 바뀌게 되고, 대부분의 경우, 번역이 다 된경우, 유전자는 비기능 단백질로 번역되게 된다. 하지만, 이는 비효율적인 방법인데, 상동 재조합은 DNA 통합(DNA integration)에서 10-2 에서 10-3 비율 밖에 차지하지 않는다. 그 보다는 더욱 많은 경우, 구조물 안에 약물을 이용한 선별 마커(drug selection marker)를 이용해 재조합 이벤트가 일어난 세포를 선별하는 방법을 이용한다.

야생형 Physcomitrella 및 녹아웃이끼: 유전자파괴를 통한 표현형의 일탈. Physcomitrella 야생형과 형질전환된 식물이 최소 Knop 배지에서 분화와 배우자 낭체를 보기 위해 배양됨. 윗줄 사진은 눈금 1mm이고 근접촬영한 아랫줄 사진은 0.5mm 눈금을 이용했다. A: 반수체(haploid) 야생형 이끼는 잎이 풍성하게 덮힌 배우자 낭체를 볼 수 있다. B-D: 다른 돌연변이
유전자가 결손된 (줄기) 세포는, 쥐의 경우에, 초기 배아에 그것을 삽입함으로써 생체에서 사용될 수 있다. 이러한 키메라 마우스가 생식 세포계에 이 유전자 변화를 가지고 있으면, 이것은 후손에게도 이어진다.
이배체에서, 대부분의 경우에 두 군데 유전자 자리를 포함하는데, 한가지 기능을 하는데 여러 유전자가 연관되는 경우, 추가적인 형질전환이나 선별과정이 목표 유전자를 녹아웃할 때까지 필요할 수 있다. 동형 녹아웃 동물을 생산하기 위해서 선발 육종(selective breeding)이 필요할 수도 있다.
****************************************************************************************************************************************
쭈욱가다가 교차가 일어나서 원따라서 가다가 다시 선에 연결된다.
그래서 플라스미드가 삽입이 된다.
독립적으로 있을 수 있지만 안으로 들어가서 삽입이 될 수 있다.
그림에서 핑크색이 안쪽으로 와야한다.
어째는 Leu2는 하나가 정상적인게 있기 때문에 selection할 수 있다.
이런 플라스미드를 에피솜이라고 부른다.
YEp13 : Yeast Episomal plasmid 라는 것이다.
플라스미드는
복제원점을 가지고 있기 때문에 cromosome과 독립적으로 복제할 수 있다.
2.5. 7.1.4 Other types of yeast cloning vector

YIp5는 반드시 삽입되어야만 하는 벡터
YRp7은 독자적으로 존재할 수 있는 벡터
YIp5
(a) pBR322를 가지고 있고 대장균에서 증식될 수 있다
위쪽을 보면 URA3를 가지고 있는데 Yeast의 크로모솜 유전자이다.
이 플라스미드를 운반체로 쓸려면 URA3- 를 써야한다.
반드시 삽입되어야만 사라지지 않고 Yeast 내에 존재할 수 있다.
YRp7
(b) pBR322가 있어서 대장균에서 재조합체를 얻을 때
TRP1은 Yeast 유전자인데 옆에 Yeast cromosome origin이 있다.
상세 설명
**************************************************************************************************************************
2.6. 1) Yeast integrative plasmids(YIps)
기본적으로 yeast gene을 가진 bacterial plasmid
URA3 gene이 삽입된 pBR322.
이 유전자는 pyrimidine nucleotide 생합성 단계 중 하나를 촉매하는 효소인 orotidine-5'-phosphate decarboxylase를 암호화하고 있으며 LEU2 와 동일한 방식으로 selectable marker로써 이용됨.
YIp 은 2㎛ circle의 어떠한 부분도 가지고 있지 않기 때문에 plasmid 처럼 복제할 수 없으며 그것의 복제는 yeast chromosome 내부로의 통합에 의존함.
2.7. 2) Yeast replicative plasmids(YRps)
Replication origin을 포함하는 chromosomal DNA 서열을 가지고 있기 때문에 독립적인 plasmid로서 복제할 수 있음.
Replication origin은 selectable marker로 이용될 수 있는 몇 개의 yeast gene과 매우 가까운 위치에 있는 것으로 알려져 있음.
Yeast gene TRP1 이 삽입된 pBR322.
Tryptophan 생합성과 관련된 이 유전자는 chromosome의 replication origin과 인접해 있다. YRp7 에 존재하는 yeast DNA 단편은 TRP1 과 그 origin을 모두 가지고 있다.
****************************************************************************************************************************************
★★★★★★★
특정 cloning 실험을 위해 어떤 유형의 yeast vector가 가장 적합한가를 결정할 때 세가지 측면에서 고려해야 한다.

1) Transformation frequency : cloned gene을 얻고자 하는 경우
2) Copy number : cloned gene 으로부터 단백질을 얻고자 하는 경우
3) Stability : 배양액 내에 오랜기간 동안 cloned gene을 보존해야 하는 경우
효율이 제일 낮고 높은데 이유가 있다.
대신 안정한것은 YIp이다.
염색체에 끼어 들어간 카피수 하나만 있다.
그래서 어떤게 목적이냐에 따라서. 안정성이 중요하다면 삽입형 플라스미드 벡터를 쓰면 된다.
Yeast에서 사용하는 세가지 플라스미드 벡터에 대해서 봤다.
2.8. 7.1.5 Artificial chromosomes can be used to clone huge pieces of DNA in yeast

Yeast 크로모솜은 박테리아 크로모솜보다 크기 때문에 용량이 더 크다.
인공염색체 벡터. 박테리아가 아닌 인공염색체
진핵섹포 염색체를 가지고 있어야한다.
Centromere : 방추사가 부착될 때 필요, 염색체를 분리할 때 필요
Replication origin이 여러개 있다. 대장균 같은 경우 Replication origin이 하나이다.
진핵세포의 origin은 여러개가 있다.
왜냐면 진핵세포의 염색체는 복제속도가 더 느리기 때문이다.
그러면서 더 크기 때문에 동시다발적으로 복제를 시작해야해서 분열속도에 복제속도를 맞춰줄 수 있다.
상세 설명
****************************************************************************************************************************************
Yeast cloning vector 의 마지막 유형은, gene cloning 에 대한 완전히 새로운 접근 방식으로써, yeast artificial chromosome을 의미하는 YAC 이다. YAC 의 개발은 진핵생물의 염색체 구조에 대한 기초적인 연구로부터 파생된 것이었다.
염색체 구조가 밝혀짐으로해서 개별적인 구성요소(centromere, telomere, origins)들이 재조합 DNA 기술에 의해 분리되고, 그 후 시험관 내에서 다시 서로 결합되어 인공염색체가 창조될 가능성이 높아졌다. 효모염색체에 존재하는 DNA분자가 수백 kb이기 때문에 인공염색체를 이용하면 어떤 다른 유형의 vector가 가질 수 있는 것보다 훨씬 더 큰 DNA단편을 cloning하는 것이 가능할 수도 있다.
****************************************************************************************************************************************
2.8.1. (a) The structure and use of a YAC vector
★★★★★★★

Yeast의 인공염색체 벡터
원래 Yeast의 플라스미드는 뒤쪽에 써주기로 했지만 앞에다가 P를 쓰고있다.
일단 제한효소 자리에 두가지를 쓰고 있다.
SUP4에 먼저 넣어준다.
어떤 기질이 있을때 red를 주지만 이 유전자를 자르면, 즉 재조합체면 이게 망가지니까 white 콜로니가 된다.
lacZ와 비슷함
이걸로만 자르는게 아니라 BamH1으로도 자르는데 이게 빠져나가게 되면 텔로미어를 양쪽에 갖게된다.
Replication origin은 TRP1 옆에 있다.
염색체 분리에 필요한 부분 CEN4
Selection marker : TRP1, URA3 : 얘내들은 안 망가진다.
숙주의 genotype을 보면 Ura3- trp1- 이다.
그래야 트립토판을 안 넣어줬을 때 숙주가 얘를 안 받아줬다면 죽어야한다.
Sup4가 없는 배지에다가 집어 넣는다.
상세 설명
****************************************************************************************************************************************
★★★★★★★
YAC vector의 전형적인 예는 pYAC3이다. pYAC3는 얼핏보면 인공염색체처럼 보이지 않지만, 자세히 관찰해보면 그것의 특징들을 가지고 있다.
pYAC3는 수많은 yeast gene이 삽입되어 있는 pBR322이다. 이들 유전자 중 두 유전자 URA3 와 TRP1 은 각각 YIp5와 YRp7에서 selectable marker 로써 이미 소개되었다. YRp7에서와 같이, TRP1 을 가지고 있는 DNA단편은 또한 replication origin을 가지고 있지만, pYAC3에서 이 단편은 CEN4 라는 서열을 포함하고 있어서 더 연장되어 있다. CEN4 는 chromosome 4의 centromere 부위로부터 얻어진 DNA이다. 그러므로 TRP1-origin-CEN4 단편은 인공염색체의 세가지 구성요소중 두가지를 가지고 있는 것이다.
세 번째 구성요소인 telomere는 TEL 이라 불리우는 두 개의 서열에 의해 제공된다. 이들은 그들 자체로 완전한 telomere서열은 아니지만, 일단 yeast 핵내에서 그들은 telomere가 형성될 seeding sequence 역할을 한다.
나머지 한 부분은 SUP4 이며 이것은 cloning 실험동안 새로운 DNA가 삽입되는 selectable marker이다.
pYAC3를 이용한 cloning 방법은 다음과 같다.
- 우선 BamHI과 SnaBI을 사용하여 double digestion시킨다.
- BamHI fragment를 제거하면 양 말단에 TEL sequence와 SnaBI site가 존재하는 두 개의 arm이 남는다.
- SnaBI(TACGTA를 인식하여 blun end를 만드는 제한효소)절단 부위처럼 blunt end를 가진 조작될 DNA를 두 arm 사이에 ligation 시켜 인공염색체를 만든다.
- Protoplast transformation 방법을 이용하여 인공염색체를 S. cerevisiae 내로 도입시킨다. 이때 사용되는 효모균주는 trp1-ura3- 인 double mutant로서 인공염색체 상의 두가지 marker에 의해 trp1+ura3+ 로 전환된다.
- 따라서 형질전환체는 정확히 제조된 인공염색체를 가진 세포만 성장할 수 있는 최소배지에 plating함으로써 선별된다.
- Vector 내에 삽입 DNA의 존재는 SUP4의 insertional inactivation을 검사항으로써 확인될 수 있다. 이것은 간단한 color test에 의해 수행된다. White colony는 recombinant이고 red colony는 아니다.

****************************************************************************************************************************************
3. 7.2 Cloning Vectors for Higher Plants

상세 설명
****************************************************************************************************************************************
숙주 생물체로써 고등식물을 이용하는 gene cloning에는 중요한 잠재적 이점이 있다. 이미 gene cloning을 통해 virus와 곤충에 저항성을 가진 실험 식물이 만들어지고 있으며, 저항성을 향상시킨 유전학적으로 조작된 tomato는 supermarket에 공급되고 있다.
고등식물을 위한 세가지 유형의 vector system이 사용되고 있다.
① Tumor-inducing(Ti) plasmid of Agrobacterium tumefaciens
Agrobacterium에 자연적으로 존재하는 plasmid에 기초한 vector
② Direct gene transfer to protoplast of plant cell
Plant cloning vector에 부착되지 않은 DNA단편을 사용하는 direct gene transfer
③ Plant virus vector
Plant virus에 기초한 vector
****************************************************************************************************************************************
식물은 작물개량에 많이 활용했다.
특히 관심있는 것은 고등식물이기 때문에 고등식물에 클로닝벡터를 어떤것으로 쓸 수 있느냐
총 세가지가 있다.
- Agrobacterium이 가지고있는 플라스미드
- 박테리아의 플라스미드에다가 우리유전자를 삽입해서 Direct gene transfer하기
- 세번째는 바이러스 벡터
3.1. 7.2.1 Agrobacterium tumefaciens-nature's smallest genetic engineer
★★★★★★★
플라스미드는 식물체 자체가 플라스미드를 가지고 있지 않으므로 식물체에 있는걸 꺼내서 쓸 수 없다.
그럼에도 블구하고 쓸 수 있는 이유는 Agrobacterium이라는 세균이 있어서
식물염색체에 recombinant 되어서 쓸 수 있다.

식물의 상처부위에 감염을 해서 엄청 세포분열 해서 혹을 만드는데 이걸 식물의 암이라고 이야기한다.
얘가 영양분 빨아먹어서 시들어서 죽게 된다.
이것을 연구하다가 뿌리혹 병을 발견했는데 병 발생기전을 분자생물학 기전으로 연구하게 된다.

3.2. Ti plasmid
Tumer indusing 이라고 한다.
사이즈가 200kb 이상으로 크다.
T-DNA의 중요한 부분 세가지
Host specific : 자연상태에서는 원래 외떡잎식물에는 감염안되고 쌍떡잎에만 감염된다. 그걸 결정하는 부위이다. 어떤 식물에 감염시킬거냐
Virulence region : T-DNA transfer에 관여
식물의 상처부위에 감염되게 되면 Virulence rigion가 T-DNA를 식물염색체에 삽입을 시킨다.
이동하는 부위 : T-DNA
나머지는 이동하지 않는다.
마치 식물의 유전자처럼 식물세포 내에서 대대로 복제되면서 발현하게 된다.
도대체 T-DNA에 뭐가있길래 아그로박테리아가 넘겼을까?
2가지 부위의 유전자가 있는데 사이토키닌과 같은 식물세포 분열을 촉진시키는 유전자가 있다.
오파인이라는 굉장히 독특한 종류의 아미노산 유도체이다. 다른 생물체들은 필요로 하지 않는다.
이것을 아그로박테리아가 필요로한다. 어떤 스토리냐
오파인같은 독특한 아미노산이 다량으로 필요하므로 이거를 식물로 하여금 만들게 한다.
식물이 많이 만들려면 공장을 지어야 하는데 마구마구 증식시켜야 해서 뿌리혹이 만들어진다.
자기가 필요로하는 유전자 산물인 오파인 계열을 대량 생산하기 위해서 식물세포에다가 유전자를 삽입하고 최초의 형질전환 세포를 엄청난 클론으로 증식시켰다.
상세 설명
****************************************************************************************************************************************
Crown gall disease를 유발시키는 능력은 이 세균내에 Ti(Tumor Inducing) plasmid의 존재와 관련이 있다. 이것은 200 kb 이상의 거대한 plasmid이며 감염과정과 관련된 많은 유전자들을 가지고 있다.
Ti plasmid의 한 가지 주목할 만한 특징은 감염 후에 그 일부가 plant chromosomal DNA 내부로 통합된다는 것이다. 이 부위를 T(transfer)-DNA라 부르며, 균주에 따라 그 크기는 15~30 kb 사이이다. 그것은 식물세포내에서 안정적인 형태로 유지되어, 염색체의 통합부위로써 딸세포로 전달된다. 그러나 가장 놀랄 만한 특징은 그 T-DNA가 식물세포내에서 발현되는 8개의 유전자를 가지고 있으며 형질전환된 세포의 암적인 특징을 담당하고 있다는 것이다. 또한 이들 유전자는 그 세균이 영양분으로써 이용하는 opine 이라 불리우는 비정상적인 화합물을 직접 합성한다. 짧게 말해서, Agrobacterium tumefaciens는 자신의 목적을 위해 식물세포를 유전학적으로 조작한다.
① Plant cell의 물리적인 상처
② phenolic compound 분비
③ A. tumefaciens가 phenolic compound인식
④ T-DNA 이동
⑤ 숙주 chromosome에 통합
⑥ Opine 합성
* T-DNA 대신 유용한 gene을 삽입(ex, human hormone gene, vitamin gene, vaccine gene)
* 단점 : 숙주가 쌍자엽 식물로 한정되어 있을 뿐 아니라 숙주 특이성이 있다.
****************************************************************************************************************************************

이 부분의 핵심 부분은 식물성장호르몬: 옥신, 사이토카인이 들어있다.
이것 때문에 바로 혹이 생기는 것이고 이게 식물의 암이다.
아그로박테리아는 이게 필요한게 아니고 다른데에서는 보기 어려운 아미노산의 유도체인 오파인이 필요하기 때문에 대량생산을 하기 위함이다.
그래서 아그로 박테리움을
자연계에 존재하는 가장 작은 Genetic Engineer라고 한다.
얘가 인간보다 빨리 형질전환 시킨 생물이다.

Ti 플라스미드가 아그로박테리움이 들어있고 대장균에다가 T-DNA일부분을 삽입을 했다. 그리고 우리의 유전자를 끼워넣었다.
아그로박테리움에 형질전환 시켜서 끼워 넣는다.
T-DNA가 상동성이 있기 때문에 recombinantion이 일어나고 T-DNA에 우리유전자가 들어간 부분이 생기게 된다.
식물에다가 상처를 내고 이 벡터를 문지르면 이 부분이 식물세포에 삽입이 된다.
그래서 우리유전자가 식물에 안정적으로 삽입이 된것이다.
근데 이게 귀찮아져서 개량을 한다.
****************************************************************************************************************************************
새로운 분자와 Ti plasmid 사이의 homology는 두 분자가 동일한 Agrobacterium tumefaciens내에 존재할 경우, 재조합에 의해 T-DNA부위 내부로 pBR plasmid가 통합될 수 있다는 것을 의미한다. 따라서 cloning될 유전자를 작은 pBR plasmid상에 특정 제한효소부위내로 삽입시키고, Ti plasmid를 가진 Agrobacterium tumefaciens안으로 도입시키면, 자연적인 재조합과정에 의해 T-DNA 내부로 새로운 유전자가 통합된다. 이 후 식물에 감염시키면 T-DNA와 함께 새로운 유전자가 식물 염색체 내부에 삽입된다.
조작되어진 Ti plasmid를 가진 Agrobacterium tumefaciens가 자연적인 방법으로 식물내로 도입된다면 crown gall을 유발시킬 세포들만 조작된 유전자를 가지게 될 것이다. 이것은 분명히 생물공학자에게 가치가 없다. 대신 식물의 모든 세포내로 새로운 유전자를 도입시키는 방법이 필요하다. 가장 간단한 방법은 성숙한 식물이 아닌 배양중인 식물세포나 protoplast에 감염시키는 것이다.
****************************************************************************************************************************************

일부로 상처를 내서 뿌리혹이 나게 한다.
이렇게 하면 식물이 나중에 결국 죽게 된다. 힘들게 지어 놓은 공장이 수명을 다하게 된다.
그래서 이걸 클론을 떨구게 되면 덩어리(캘러스)를 만들게 되고
분화 배지에다가 옮기면 분화가 돼서 싹이 나면서 식물이 자라게 된다.
이 식물의 경우에는 최초의 생물에서 모든세포가 나왔기 때문에 이 식물의 모든 세포는 형질전환이 되어있는 것이다.
오리지날은 암을 만들어서 죽기 때문에 이것을 생각한 것이다.

T-DNA 양쪽 끝에 반복서열이 있다. 25bp 짜리


양끝을 인식할수 있다는게 25bp 이다.
25bp만 떼어서 다른 유전자에 부착시키면?
25bp만 있으면 된다라는 것.
그것을 착안해서 만든 것이 pBIN19 이다.
10kb로 줄인 것

이걸 아그로 박테리아에 넣었을 때 노란 파랑 부분만 들어간다는 의미이다.
식물에다가 완성된 박테리아에 lacZ에 우리유전자가 삽입된걸 확인 한다.
이거를 아그로 벡티리움에 넣는 것이다.
온전한 플라스미드 Ti 플라스미드를 가지고 있으면 비룰런스가 인식해서 넣어주게 된다.
박테리아에서 이 재조합체를 만들어야 하기 때문에 이 플라스미드는 박테리아에서 필요한 두개의 셀렉션 마커를 갖는다.
kanR 를 쓴 이유는 이 캐너마이신이라는 항생제는 특이하개도 대장균도 죽이지만 식물셀도 죽인다. 무슨말이냐면 항생제 마커가 일석이조가 되는 것이다.
대장균에서도, 식물세포에도 셀렉션 마커로 쓰일 수 있다는 것이다.
T-DNA일부를 갖게해서 통합된다음에 들어갈 필요는없고 같은 공간에 있어도 된다.
아그로박테리움에 넣어야 나머지 ti 플라스미드가 넘겨준다.

결국 외떡잎식물 말고도 쌍떡잎에도 쓸 수 있게 만들었다.
3.3. 7.2.2 Cloning gene in plants by direct gene transfer

두번째는 그냥 박테리아의 플라스미드를 쓰는 것이다.
이걸 식물에다가 다이렉트로 넣어준다 유전자 총을 쏘든 뭘하든
그러면 supercoil을 갖기 때문에 식물의 염색체에 통합된다. 효율은 그렇게 높진 않지만.
이것은 아무 부위나 랜덤하게 삽입 될 수 있다. 원리는 모른다.
효율은 아그로박테리움보다 낮다.
상세 설명
****************************************************************************************************************************************
Selectable marker와 조작될 유전자를 가진 supercoiled plasmid DNA를 어떻게 식물세포 내부로 도입시킬 수 있는가?
3.3.1. 1) Original method
Polyethylene glycol, polymeric negatively-charged compound로 구성된 점착성 용액에 재현탁된 protoplast를 이용하는 방법으로, protoplast표면에 DNA를 침전시키고 endocytosis에 의해 uptake를 유도시킨다고 생각되고 있다.
3.3.2. 2) More efficient method
Electroporation, microinjection, 그리고 DNA-containing liposome을 이용한 융합법 등이 최근 이용되고 있다. 처리 후에는 세포벽을 재생성시키기 위해 protoplast를 며칠 간 용액속에 방치시킨다. 그 다음에 형질전환체를 동정하기 위해 선택배지에 plating하고 callus를 완전한 식물이 성장될 수 있는 배지로 옮긴다.
****************************************************************************************************************************************

이렇게 다이렉트하게 넣으려면 식물의 벽을 제거하고 프로토플라스트를 만들어서 넣어야한다.
DNA가 표면에 붙고 endocytosis :세포막함입을 통해서 DNA가 들어간다.
여기서 아그로박테리움을 이용하면 세포벽을 벗길 필요가 없이 알아서 감염을 하기 때문이다.
다이렉트하게 넣으려면 세포벽을 제거하고 넣어야한다. 식물의 경우 Regeneration이 되려면

식물에서 또 유전공학적으로 관심 있는 것은
식물하면 엽록체가 중요하므로 여기에 넣고 싶은 경우가 많다.
Chloroplast 타겟팅을 위해서 쓸 수가 있다.
Chloroplast에 DNA가 들어가면 homologous recombination에 의해서 들어간다.
유전자 총 : Biolistics
PEG 로 들어갈 수 있지만 어렵다.
장점 : 세포당 핵은 하나고 지놈은 카피수가 한두개지만 클로로플라스트 안에는 지놈이 수백개가 있다. 또 세포당 염색체가 많기 때문에 수만 카피가 일어날수 있기 때문에 카피가 많아진다.
Selection 할 때에는 오랜기간동안 해야한다. 모든 지놈에 들어가야 하기 때문에.
1. 아그로박테리아
2. 그냥 떄려넣기
3. 바이러스
이제 바이러스 보자
3.4. 7.2.3 Attempts to use plant viruses as cloning vectors

쉽게 우리유전자 삽입할 수 있다.
여기서 문제점은 식물의 바이러스의 대부분은 RNA바이러스이다.
그래서 RNA에 우리유전자를 넣으면 전부 double strand를 활용하는 것이므로 쓸 수 있는 바이러스가 상당히 제한되어 있다.
CaMV : size 제한이 있다. 람다 처럼 사이즈 제한이 있다. 람다처럼 불필요한 부분을 삭제해야한다.
너무 삭제하면 바이러스의 기능을 잃어버려서 헬퍼, CaMV를 넣어준다.
M13페이즈를 너무 적게 남겨서 헬퍼가 필요한 것처럼
Geminivirus : 외떡잎식물에 쓸 수 있는 바이러스라서.
이것의 단점이 뭐냐면 Rearrangement가 많이 일어나서 바이러스가 변이되기 쉽다.
이것의 아주 중요한 응용 : VIGS 라는 테크닉이다.
상세 설명
****************************************************************************************************************************************
대부분의 식물들은 virus에 의해 감염된다. 그렇다면 식물에서 유전자를 조작하기 위해 virus가 이용될 수 있을까? 한가지 문제점은 주요한 식물 virus가 RNA genome을 가지고 있다는 점이다. RNA는 조작하기가 어렵기 때문에 RNA virus는 cloning vector로써 유용하지 못하다. DNA virus중 단지 두 종류가 고등식물에 감염하는 것으로 알려져 있다(caulimoviruses and geminiviruses).
3.4.1. 1) Caulimovirus vector
: 두 가지 단점을 가지고 있다.
① Size limitation
Non-essential section을 제거시킨 후에도 삽입될 수 있는 DNA크기는 여전히 매우 제한적이다(0.2~0.3kb)
② Narrow host range
숙주 범위가 매우 좁아서 단지 순무, 양배추, 꽃양배추에만 조작실험이 가능하다.
3.4.2. 2) Geminivirus
옥수수와 밀 같은 중요한 작물에 자연적으로 감염하기 때문에 얼핏보면 유용할 것 같지만, infection cycle동안에 genome의 재배열과 결손이 일어나기 때문에 삽입된 DNA에 어떤 추가적인 DNA가 뒤섞일 수 있다. 또한 이 virus는 작물들에게 감염으로 인한 피해를 일으킬 수 있기 때문에 geminivirus cloning vector의 사용은, 숙주로부터 빠져나온 vector의 위험성과 자연상의 식물군집에 감염되는 것을 피하기 위해 엄격한 통제하에서 수행되어야 한다.
****************************************************************************************************************************************

바이러스에 의해서 유도되는 유전자 친목화.
유전자를 망가뜨린다. Gene silencing 기법
우연히 뭘 삽입해서 망가뜨릴 수 있겠지만
또 한가지 방법은 그 유전자가 발현될 때 mRNA를 무력화 시키는 방법을 쓰면 된다.
메신저 RNA의 상보적인,
제미니 바이러스에 유전자 일부를 집어 넣으면 이걸 식물에 도입하면
잉 유전자 부분이 전사가돼서 mRNA가 나온다. 이게 siRNA 역할을 한다.
이 RNA

얘에서 발현되는 메신저 RNA를 무력화 시킨다.
RNA간섭현상을 일으키는 작은 것 : siRNA
세포내에 이런 기작으로 간섭현상으로 특정 기작을 조정하는 것은 microRNA이다.
맨처음 이스트
식물 벡터
다음시간 동물
'🧬 Bio > 유전공학' 카테고리의 다른 글
| 유전공학 8장 (2), 9장 - 유전자 및 유전체 구조 연구 (0) | 2020.04.16 |
|---|---|
| 유전공학 7장 (2), 8장 - 특정 유전자의 클론을 얻는 방법 (0) | 2020.04.13 |
| 유전공학 6장 - 대장균(E. coli) 용도의 클로닝 벡터 (0) | 2020.04.06 |
| 유전공학 5장 - Introduction of DNA into living cells (0) | 2020.04.02 |
| 유전공학 4장 - Manipulation of purified DNA (0) | 2020.03.30 |