
박테리아의 개량 벡터
- pBR322
- Copy number : 15
- 크기 < 10kb
- Selection marker : ampR, tetR
- pBR327
- Copy number : 30-45
- Conjugative ability를 못하게 만듦 (항생제 내성을 갖는 슈퍼박테리아가 나올 수 있어서)
- biological containment : 생화학적 제약
- pUC8
- Copy number : 500-700
- Selection marker : ampR, lacZ’
- lacZ를 써서 one-step selection이 가능하다. White Colony 추출
- Mutiple cloning site : 여러개의 제한효소 자리가 있다.
- T7 & SP6 promoter
- T7과 SP6는 박테리오페이즈이다.
- 자기의 RNA polymerase를 활용하여 자기 유전자를 많이 만든다(전사한다).
- M13 Vector
- M13mp1 : lacZ’ gene 이 삽입된 벡터
- M13mp2 : EcoR1 gene 이 삽입된 벡터
- M13mp7 : polylinker가 합성되어 cloned DNA가 쉽게 복구될 수 있는 벡터
- M13 vector는 size limitation이 있다
- 이중가닥의 M13 DNA를 단일가닥으로 변환시키는 효소에 의해 인식되는 Signal sequence 존재
- Helper phage : 복제효소와 phage coat protein을 공급한다.
- λ phage
- 크기가 커서 외부 DNA의 삽입이 어렵다 : Non-essential region 제거
- lysogenic cycle 관련 부위를 제거 -> lytic cycle만 갖게 한다.
- 제한효소 부위가 여러군데 있으므로 이 부위를 제거한다.
- 크기가 커서 외부 DNA의 삽입이 어렵다 : Non-essential region 제거
- cosmid : 가장 정교한 유형의 λ-based vector
- phage DNA + plasmid DNA
- λ DNA를 phage protein coat안으로 packaging 시키는 효소가 기능하기 위해 cos site만을 필요로 하는 특성을 이용
- cos site를 가지고 있는 plasmid
- λ gene 이 결손되어서 plaque 형성 불가, colony 형성
- phage로 packaging 될 수 있으나 lytic cycle에 관련된 부위가 존재하지 않기 때문에 plaque를 형성할 수 없다.
- size로 알 수 있기 때문에 selection이 필요가 없다.
6. 대장균(E. coli) 용도의 클로닝 벡터

"p" = 이것이 plasmid임을 가르킨다.
"BR" = vector를 개발한 laboratory를 말한다(BR은 pBR322를 개발한 두 연구자 Bolivar와 Rodriguez)
"322" = 같은 실험실에서 개발한 다른 plasmid와 구별하기 위한 번호
multi-enzyme site : 여러 제한효소 부위의 집합 부위; also called multi-cloning site, restriction site cluster
pBR322

엠피실린과 망가진 BamH1 이어야 우리가 원하는 것.
운반체 자체가 너무 크면 pipet으로 옮길 때 미세한 힘으로 인해 DNA가 잘라진다.
copy number 15개 정도되고 플라스미드 amplify로 인해서 증폭이 될 수 있다.
사이즈가 10kb가 넘으면 쓰기 힘들다.
플라스미드는 Selection marker가 2개가 있어야 한다. : ampR, tetR
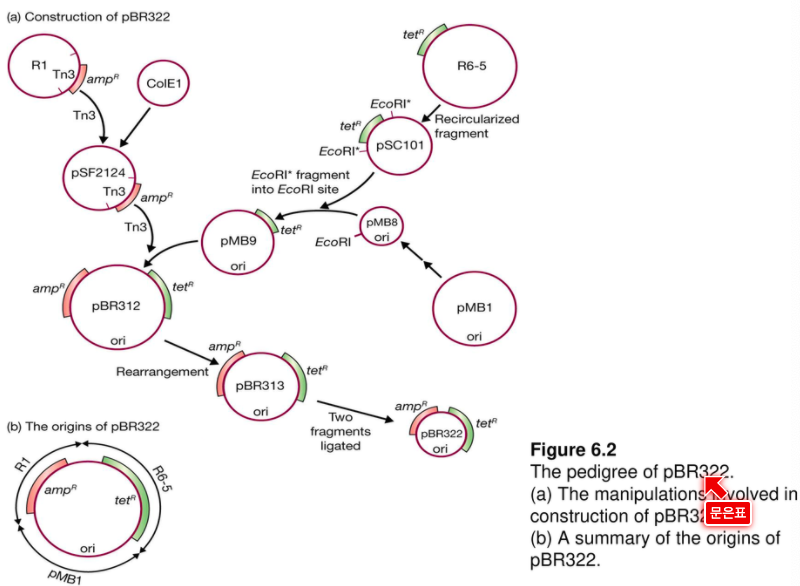
**** 차이점 기억해라
pBR327

1. copy number 억제를 덜 받게 된다.
2. conjugation을 못하도록 삭제를 해버림 (323는 가능) -> 수퍼박테리아가 나올 수 있기 때문에 ****
이게 왜
갖고 있는 것이 conjugation이면 물리적인 접촉으로 플라스미드를 복제해서 넣어준다.
유전물질을 수평적으로 전파할 수 있다.
그런데 옆에있는 애한테도 전달할 수 있다.
이런애들이 자연계로 나가면 접합에 의해서 형질전환체가 전달할 수 있다.
이때 항생제내성유전자를 마구마구 전파시키면 수퍼 박테리아가 나오고
인간에게 병을 일으키는 세균이 되면 이건 항생제로 죽일 수 없는 것이 된다.
수평적 전달을 차단해야하기 때문에 생물학적 제약을 둔다. → biological containment
상세 설명
****************************************************************************************************************************************
pBR327 - a higher copy number plasmid
pBR327은 pBR322에서 1089 bp를 제거함으로써 만들어졌다. 그 결과 pBR327은 두 가지 중요한 측면에서 pBR322와 차이점이 있다.
- pBR327은 pBR322보다 copy number가 많다(30~45분자/cell). pBR327의 이러한 higher copy number 때문에, 그 실험의 목적이 cloned gene의 기능을 연구하는 경우에 이 vector가 더욱 적합하다.
- pBR327은 pBR322의 접합능력이 파괴되어 있다. 이것은 부주위한 분자 생물학자들의 실수로, recombinant pBR327이 시험관 밖으로 유출되어 bacterial colony가 형성될 가능성을 제거시키기 위한 biological containment를 위해 중요하다.
****************************************************************************************************************************************
pUC8

1. copy number control을 조절해서 엄청 많이 나오게 한다.
2. lacZ 를 써서 one-step selection 가능 : amp, lacZ
lacZ' 이 망가진 것을 고르면 된다. 하얀 콜로니
3. pBR322 나 327는 넣을 자리가 많지 않았는데 MCS라고 해서 여러개의 제한효소 자리가 있다. ****
상세 설명
****************************************************************************************************************************************
pUC8 - a Lac selection plasmid
pUC8은 pBR322로부터 유도되었으며 ampicillin resistance gene의 서열을 변화시켜 restriction site가 없도록 하였다. 모든 cloning site는 lacZ' gene의 짧은 부위에 밀집되어 있다.
pUC8은 세 가지 중요한 장점을 가지고 있다.
- replication origin내에 우연한 돌연변이에 의해 500~700 copy number를 가지고 있다. 이것은 recombinant pUC8 plasmid를 가진 E. coli 로부터 얻을 수 있는 cloned DNA의 수율이 매우 높다는 것이다.
- single-step만으로 recombinant cell을 동정할 수 있다. 이는 pUC8이 pBR322나 pBR327이 요구하는 시간의 절반만 필요로 한다는 것이다.
- restriction site의 밀집 = Multiple cloning site 존재
DNA 단편이 두 개의 서로 다른 sticky end(이를 테면 한쪽은 EcoRI 그리고 다른 한쪽은 BamHI)를 가지도록 하여 linker 부착 같은 추가적인 조작이 필요치 않다. 게다가 이 vector의 restriction site cluster는 M13mp series의 cluster와 동일하여 pUC series에 cloning된 DNA는 M13mp로 직접 이동될 수 있기 때문에 DNA sequencing 또는 in vitro mutagenesis에 의해 분석될 수 있다.
****************************************************************************************************************************************
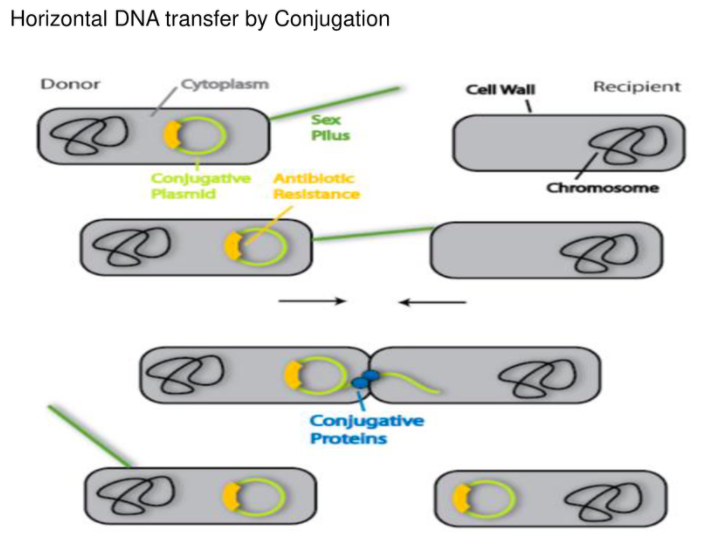
접합성을 빼버렸기 때문에 항생제가 없는 자연계로의 수평적 전파를 봉쇄한다.
생물학적 제한
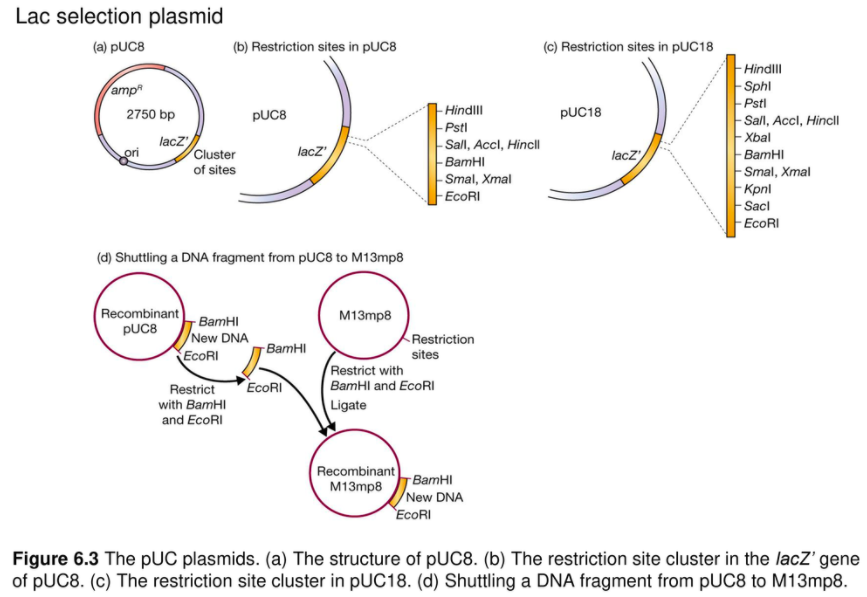
lacZ' 안에 다양한 제한효소를 넣을 수 있는 linker를 사용함으로써 다양한 제한효소를 쓸 수 있게 한다.
T7 & SP6 promoter

플라스미드 벡터 중 기억해야 할 것 중 하나 ****
lacZ ' 에 우리 유전자를 삽입하고 여기 들어간 유전자는 T7 promoter가 전사체를 만들 수 있게 한다.
# 전사체 : 전사체는 발현된 모든 RNA의 총합을 뜻한다
T7 RNA polymerase를 넣어주면 실험관 내에서 프로모터를 인지해서 전사를 한다.
1-2 mg 은 엄청난 양의 전사량이다.
T7 과 SP6는 박테리오페이즈 이다.
원래는 대장균의 전사효소를 써서 나눠가져야 하지만
T7와 SP6는 자기 자신의 프로모터를 가지고 있어서 대장균 유전자의 프로모터와는 다르다.
자기의 RNA polymerase를 활용해서 자기 자신의 유전자만 전사하기 때문에 굉장히 많이 전사할 수 있다.
그래서 껍데기 단백질도 만들고 자기 유전자를 많이 만든다.
M13 vertor(phage)
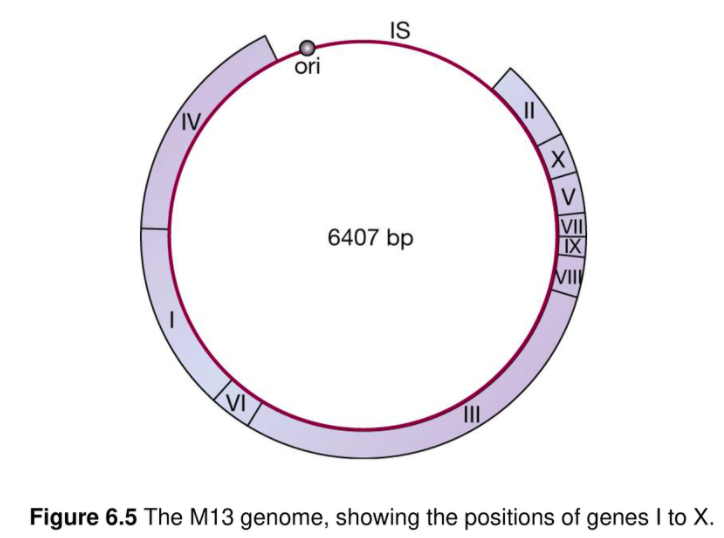
박테리오 페이즈 타입의 운반체.
람다 와 M13
개량을 안하면 활용도가 떨이지기 때문에 이것도 개량해야한다.
M13은 굉장히 작다.
M13은 ssDNA genome이다. 람다는 dsDNA genome이다.
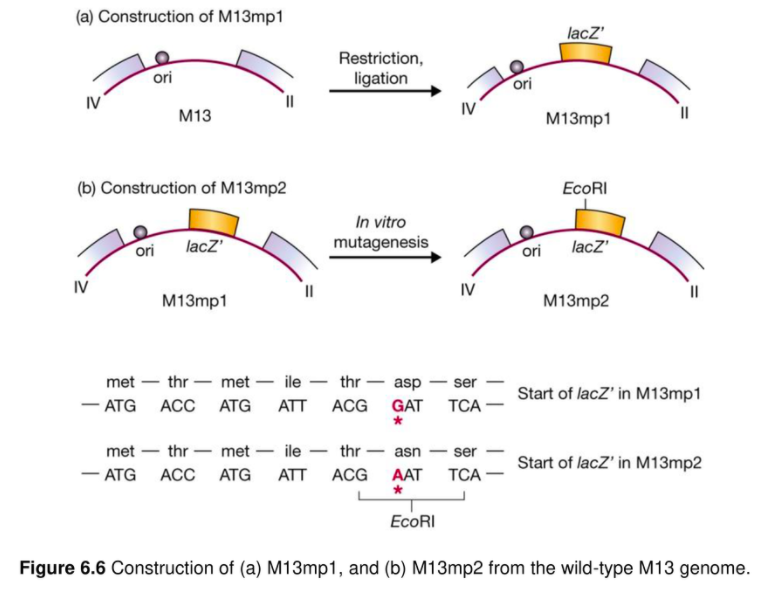
M13의 개량
selection marker로 lacZ' 을 넣어준다.
플라크가 생겼나 안생겼나로 구분할 수 있기 때문에 2스텝 안해도 됨
상세 설명
****************************************************************************************************************************************
Development of the cloning vector M13mp2
M13 cloning vector의 제조에서 첫 번째 단계는 intergenic sequence에 lacZ' gene을 삽입시키는 것이다. 이것이 M13mp1 이다. M13mp1은 lacZ' gene내에 어떤 restriction site도 가지고 있지 않다. 하지만 hexanucleotide GGATTC를 가지고 있으며 in vitro mutagenesis에 의해 single nucleotide를 변화시킴으로써 EcoRI site인 GAATTC를 만들수 있다. 이것이 M13mp2 이다. M13mp2는 조금 변화된 lacZ' gene을 가지게 되었지만 β-galactosidase는 정상적인 기능을 수행한다.
****************************************************************************************************************************************
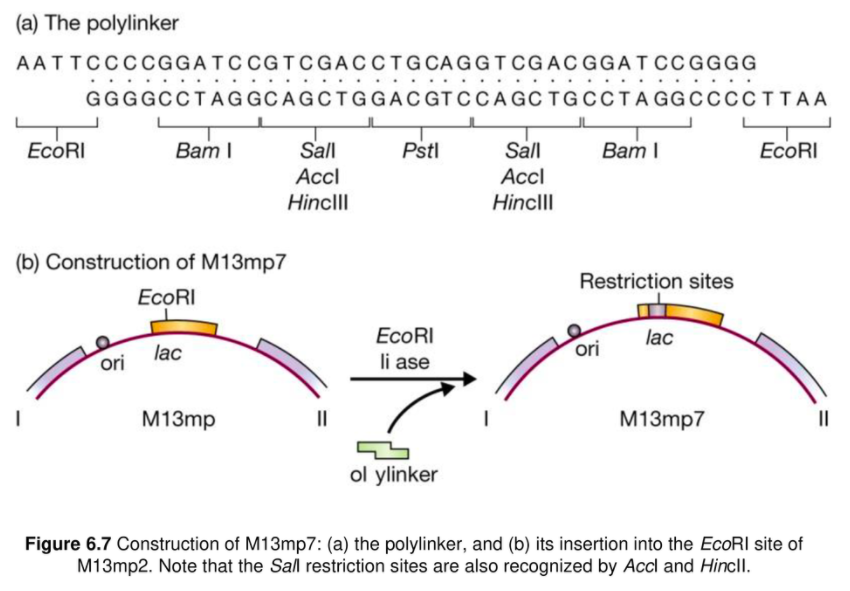
DNA조각
상세 설명
****************************************************************************************************************************************
M13mp7 - symmetrical cloning sites
M13 vector 개발에서 다음 단계는 lacZ' gene 내에 추가적인 restriction site를 삽입시키는 것이다. 우선 일련의 restriction site로 구성되어 있으면서 EcoRI sticky end를 가진 polylinker를 합성한다. 이 polylinker를 M13mp2의 EcoRI site에 삽입시키면 M13mp7이 된다. 이 polylinker는 lacZ' gene을 전체적으로 파괴시키지 않도록 구상된 것으로 reading frame이 유지되며, 변하긴 했지만 기능적인 β-galactosidase는 여전히 생산된다.
Symmetrical cloning site를 가진 M13mp7의 큰 장점은 BamHI, SalI 또는 PstI site에 삽입된 DNA를 EcoRI을 사용하여 recombinant molecule로부터 excision시킬 수 있다는 점이다. 이렇게 쉽게 cloned DNA를 복구시킬 수 있는 vector는 매우 드물다.
****************************************************************************************************************************************

어떻게 단일가닥을 이용하냐
M13의 경우는 단일가닥을 주입하면 맨 처음 일어나는 일이 (+) 가닥에 상보적인 (-) 가닥을 만들어서
ds genome으로 만든다.
복제를 위한 중간 산물이다 RS
M13은 자기가 가지고있는 (+) 가닥에 상보적인 (-) 가닥을 만들어서 Double strand처럼 존재한다.
M13 region 부위가
복제단백질을
줄줄이 사탕처럼 복제를 하게된다. 단일가닥을 만들어낼 수 있다.
단백질도 만들어지면 M13 패키지가 만들어진다.
헬퍼페이즈 (정상적인 M13) : 복제효소와 phage coat protein을 공급한다.
그래서 융합벡터가 만들어져서 DNA복제함
상세 설명
****************************************************************************************************************************************
Phage DNA분자는 일반적으로 복제를 위해 필수적인 몇 개의 유전자로 구성되어 있으며 이들 유전자 중 어떤 것의 결실은 복제능력을 손상시키거나 파괴시킨다. 따라서 phage DNA분자를 변화시키는 데는 많은 제약이 따르고 일반적으로 phage cloning vector들은 모체분자와 아주 작은 차이점만을 가지고 있다.
* phage DNA는 대부분 필수적인 gene으로 구성되어 있기 때문에 외부 DNA를 삽입시키는데 어려움이 있다.
* M13 DNA는 ①size가 작고(6.4kb), ②오염없이 DNA를 얻을 수 있고, ③ssDNA와 ④dsDNA를 얻을 수 있다는 장점이 있다.
M13 vector의 한가지 단점은 size limitation이다. 이를 해결하기 위해 plasmid DNA와 M13 genome 일부를 결합시킨 새로운 vector(phagemid)가 개발 되었다.
M13 genome 중 1300 bp 단편을 pUC8에 삽입시켜 만들었다. 삽입된 M13 DNA는 새로운 phage가 방출되기 전에 정상적인 이중가닥의 M13 DNA를 단일가닥으로 변환시키는 효소에 의해 인식되는 signal sequence를 포함하고 있다. 따라서 pEMBL8은 단일 가닥의 DNA로 변환되어 defective phage particle로서 방출된다. 실험을 위해 필요한 것은 복제효소와 phage coat protein을 공급하는 helper phage역할을 할 정상적인 M13과 이 phage에 감염될 E. coli 이다.
****************************************************************************************************************************************
λ phage

람다를 봐보자.
람다의 경우 genome size가 49kb다. 좀 크다.
그래서 이 람다를 벡터로 사용하기에는 너무 크다. 그래서 사이즈를 줄여야 한다.
사이즈가 크면 DNA를 복제한 다음에 잘라서 껍데기 단백질로 패키징 할 때
자기 genome 사이즈와 같은 것만 패키징한다.
5% 정도만 허용한다. 52kb까지 정도만.
상세 설명
****************************************************************************************************************************************
Cloning Vectors Based on λ Bacteriophage
phage λ 가 lysogenic cycle 상태일 경우 vector 개발에 필요가 없기 때문에 lytic cycle로 induction시켜야 한다.
* λ DNA를 vector로 개발하는데 문제점
①size가 커서 3 kb 이상의 외부 DNA의 삽입이 어렵다(size limitation)
해결 : lysogenic cycle과 관련된 부위(15 kb)를 제거한다 → 18 kb 까지 삽입이 가능해진다.
②size가 커서 제한효소 부위가 여러군데 존재한다(multiple restriction site)
ex)EcoR I site = 5 sites
해결 : 제한 효소 부위를 제거한다.
****************************************************************************************************************************************

껍데기 만드는 유전자이다.
이 부위는 유전공학에서는 필요하지 않는 부분이다.
이부분을 제거하면 18Kb 정도를 넣을 수 있게 된다.
Non-essential region(15kb)을 제거한다. 따라서 18kb의 new DNA 삽입이 가능해진 것이다.
Non-essential region은 대부분 integration과 excision에 관련된 유전자들이며 따라서 lytic cycle만을 가지게 된다
(1) 자연선택으로 인한 사이즈 줄이기

제한효소자리가 돌연변이에 의해서 두개나 세개가 없어졌다.
이걸 E.coli에 집어 넣으면 다 무력화 시킨다.
이것을 피한 것은 살아남는다.
그러면 EcoR1이 없어진 걸 찾을 수 있다.
이런 애들은 적자생존이 돼서 EcoR1이 없는 거만 선택이 된다.
이때는 기술이 없기 때문에 자연선택으로 만든것이다.
상세 설명
****************************************************************************************************************************************
method I(natural selection) : E. coli에 λ DNA를 infection시킨다.
그러면 제한효소에 의해 λ DNA가 파괴되다가
돌연변이가 생겨 생존하는 것이 나타난다.
↓
계속
↓
EcoR I site가 제거된 λ DNA를 얻을 수 있다.
<주의>vector로서 유용하기 위해선 제한효소부위가 전혀 없는 것 보다는 1개 있는 것이 좋다.
****************************************************************************************************************************************
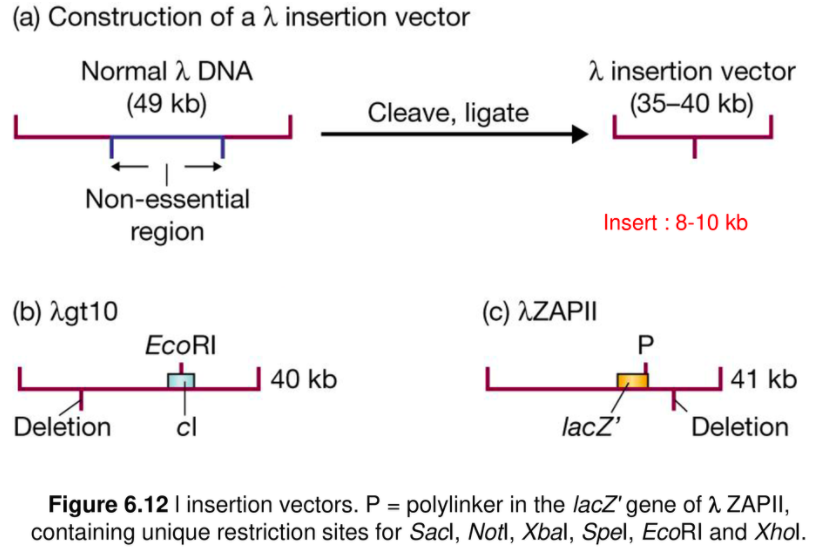
35-40kb 만을 남기면 자기 genome 보다는 작아졌지만 이런애들은 건진다. 패키징 한다.
얘내를 증식해서 우리의 DNA를 넣는다 8-10kb 정도
이렇게해서 만들어진게 λgt10이나 λZAPll이다.
(b) clear 플라크를 만든다. EcoR1 : 제한효소
(c) blue 플라크 버리고 white 플라크를 취하면 이 재조합 genome을 가지는 람다 집합체를 얻을 수 있다.
상세 설명
**************************************************************************************************************************
Insertion and replacement vectors
일단 위의 두 문제가 해결된 후에, 제조될 수 있는 두 종류의 vector는 λ-insertion과 λ-replacement(or substitution) vector이다.
(a) Insertion vectors
Non-essential region이 제거된 insertion vector를 다시 ligation시킨다. Insertion vector는 새로운 DNA가 삽입될 수 있는 독특한 restriction site를 적어도 하나 가지고 있다. 삽입될 수 있는 DNA단편의 크기는 제거된 non-essential region의 크기에 달려 있다.
1) λgt10
8kb의 새로운 DNA가 삽입될 수 있으며 cI gene내에 하나의 EcoRI site가 존재한다.
2) λZAPII
lacZ' gene내에 6개의 restriction site를 가진 polylinker가 존재하며, 10kb의 새로운 DNA 가 삽입될 수 있다.
(b) Replacement vectors
제한효소에 대한 인식부위가 두 군데 존재한다. 이들 인식부위는 새로운 DNA에 의해 대체되는 DNA부위의 측면에 존재한다. Replacement vector는 일반적으로 insertion vector보다 더 큰 DNA 단편을 삽입시킬 수 있도록 design되었다.
1) λWES.λB'
Replacement fragment 측면에 두 개의 EcoRI site 존재.
Recombinant selection은 size에 근거함.
2) λEMBL4
20kb의 새로운 DNA 삽입 가능
EcoRI, BamHI, SalI site가 쌍으로 존재
Recombinant selection : size, Spi phenotype
****************************************************************************************************************************************

왕창 잘라낸다.
EcoR1 BamH1 잘라서 우리 DNA를 넣을 수 있다. 20kb 이상 짜리를 넣을 수 있다
이게 λ replacement vector 이다.
상세 설명
****************************************************************************************************************************************
Cloning experiments with λ insertion or replacement vectors
1) Plasmid vector와 동일한 방법
이 실험을 위해선 λ vector의 cos site가 수소결합함으로써 원형을 이루어야 한다.
2) Left arm과 right arm을 순수분리 시킨 후 cloning될 DNA와 혼합한다. 그러면 left arm-DNA-right arm을 가진 concatamer가 만들어진다. in vitro packaging 시킨 다음 host cell에 infection 시킨다.
****************************************************************************************************************************************
Cosmid
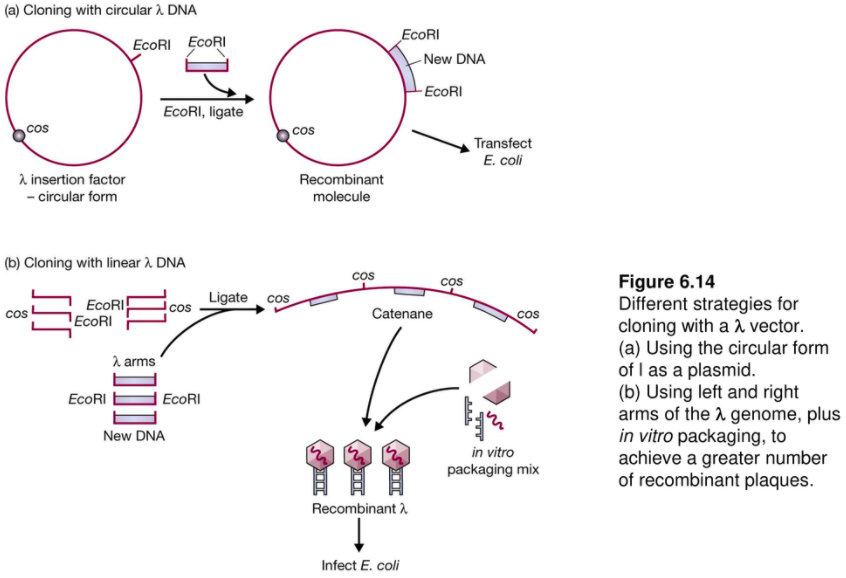
cos site는 점착성이기 때문에
cos - cos 사이를 잘라서 껍데기 단백질을 조립할 수 있게.
상세 설명
**************************************************************************************************************************
Very large DNA fragments can be cloned using a cosmid
최종적이면서 가장 정교한 유형의 λ-based vector는 cosmid이다. cosmid는 phage DNA와 bacterial plasmid를 혼성화시킨 것으로 λ DNA를 phage protein coat안으로 packaging시키는 효소가 기능하기 위해 cos site만을 필요로 한다는 사실에 입각하여 구상되었다.
in vitro packaging 반응은 λ genome뿐만 아니라 37~52 kb 의 DNA로 나누어지는 cos site를 가진 어떤 DNA에서도 일어난다. cosmid는 기본적으로 cos site를 가지고 있는 plasmid이다. 이것은 또한 ampicillin resistance gene같은 selectable marker와 plasmid의 replication origin을 필요로 하며 cosmid는 모든 λ gene이 결손되어 있기 때문에 plaque를 형성할 수 없다. 대신 selective에서 colony가 형성된다.
- cosmid = phage DNA + plasmid DNA
- 존재항목 : cos site, selectable marker, replication origin of plasmid.
- 특징 : phage로 packaging 될 수 있으나 lytic cycle에 관련된 부위가 존재하지 않기 때문에 plaque를 형성할 수 없다.
- 장점 : 40 kb 까지 외부 DNA를 삽입시킬 수 있다.
****************************************************************************************************************************************

cos site와 플라스미드를 융합한것, 아까는 m13과 플라스미드를 얻을걸 봤다.
cos site가 핵심.
람다 페이즈와 플라스미드의 융합벡터 : λphage + plasmid
BamH1으로 연다.
우리 유전자가 삽입된건지 모른다.
그런데 자르면 각각 나온다.
여기에다가 우리의 DNA 조각을 BamH1으로 잘라서 넣는다.
그러면 DNA가 쏙쏙 들어간다.
만약 BamH1으로 자른게 안들어갔다 그러면 너무 짧아서 패키징이 안된 것이다.
사이즈로 알 수 있어서 selection이 필요가 없다.
배지에 깔면 콜로니를 만든다. 플라크가 만들어지는게 아니라.
플라스미드로 자라기 때문에 콜로니가 형성이된다.
중간에만 람다 페이즈를 사용하는 것이다. 플라스미드로 증식한다.
상세 설명
< cosmid를 이용한 실험 방법 >
- 제한효소(BamH1)를 사용하여 cosmid를 개방시키고 새로운 DNA단편을 삽입시킨다.
- Ligation을 통해 concatamer를 형성시킨다.
(concatamer : DNA분자가 반복 배열되어 있는 집합체. 파지가 증식하는 과정에서, 세포 내부에 DNA가 이어져서 형성된다) - in vitro packaging에 의해 cos site가 절단되고 recombinant cosmid가 mature phage particle 안으로 들어간다.
- 이 λ phage를 E. coli culture에 infection시킨다.
- 물론 plaque가 형성되지는 않지만 대신 selective medium에 plating하면 recombinant colony가 형성된다.

Cosmid는 페이즈의 장점 따옴
replication origin은 깨고 나오지 않는다.
그래서 얻는 것은 깨고 죽는 플라크가 아니라 증식되는 콜로니로 얻는다.
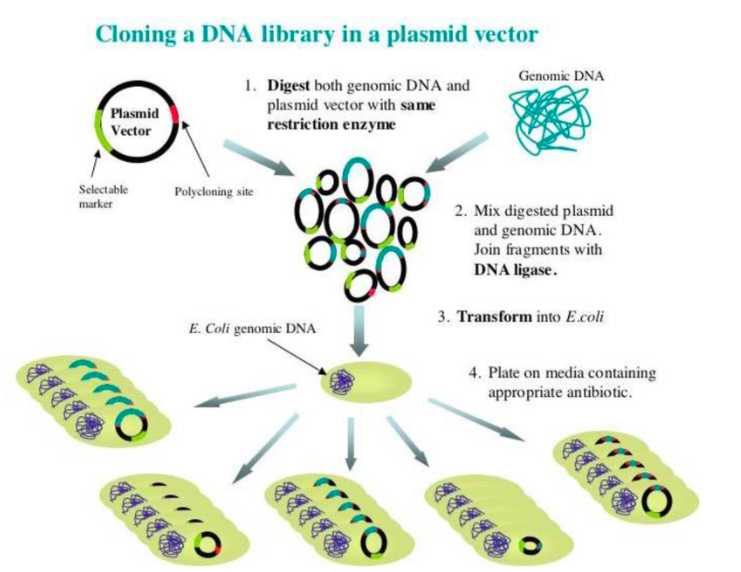
컬렉션을 얻으면 이게 라이브러리이다.
좋은 라이브러리는 그 개체가 가지고 있는 모든 DNA를 카바할 수 있게 된다.
하나를 골라낼 때 확실하게 한번 골라내고 싶다. 그러면 라이브러리를 몇개를 뒤져봐야 하냐 를 통계학적으로 계산해야 한다.


17kb로 자르면 한세트 안에 클론의 개수가 증가한다.
genome size가 같다고 했을 때 크게 자르느냐 작게 자르냐에 따라서 클론의 개수가 작아진다.
그래서 어떤 라이브러리에서 몇개를 뒤져야 내 유전자가 95%로 나오느냐
직관적으로 알 수 있다.
f 는 genome size에 대한 클론 DNA(작게잘랐느냐 크게 잘랐느냐)

라이브러리 클론에 81만개를 깔면 내가 원하는 1개가 있을 확률이 99퍼센트.
한번 81만개
건초더미에서 하나를 찾는 것 이기 때문에 큼직큼직하게 자르면 그나마 고를 수 있고 그거를 세분화해서 고를 수 있다.
공식 외울필욘 없다. 공식 주고 원리를 설명할 수만 있으면 된다.
56만4천개를 깔아야 얻는다.
크게 자르면 27만4천개를 깔아서 얻을 수 있다.

용량 기억해

대용량 벡터.
플라스미드 페이즈 코스미드는 다 다뤘고
BAC은 300플라스미드
P1 버리고 그밑에도 버리고
PAC 이정도
대장균이 아닌 다른 박테리아의 운반체에 대해서
6장에서는 박테리아의 개량벡터
5장은 플라스미드의 개량벡터
7장은 진핵세포의 개량벡터
'🧬 Bio > 유전공학' 카테고리의 다른 글
| 유전공학 7장 (2), 8장 - 특정 유전자의 클론을 얻는 방법 (0) | 2020.04.13 |
|---|---|
| 유전공학 7장 - 대장균 이외의 다른 생물체 용도의 클로닝 벡터 (0) | 2020.04.09 |
| 유전공학 5장 - Introduction of DNA into living cells (0) | 2020.04.02 |
| 유전공학 4장 - Manipulation of purified DNA (0) | 2020.03.30 |
| 유전공학 4장 - Electrophoresis (0) | 2020.03.26 |