

유전체 구조
염색질(Chromatin) : DNA + 단백질 결합체
히스톤 : 결합단백질
비히스톤 : DNA 복제, 전사 담당
뉴클레오솜 : DNA + 히스톤
염색체는 원형 혹은 선형일 수 있다.

Topoisomerase : 원형 염색체가 복제된 후 딸 분자를 분리하는 역할
모든 세포는 특정수의 염색체를 가진다.
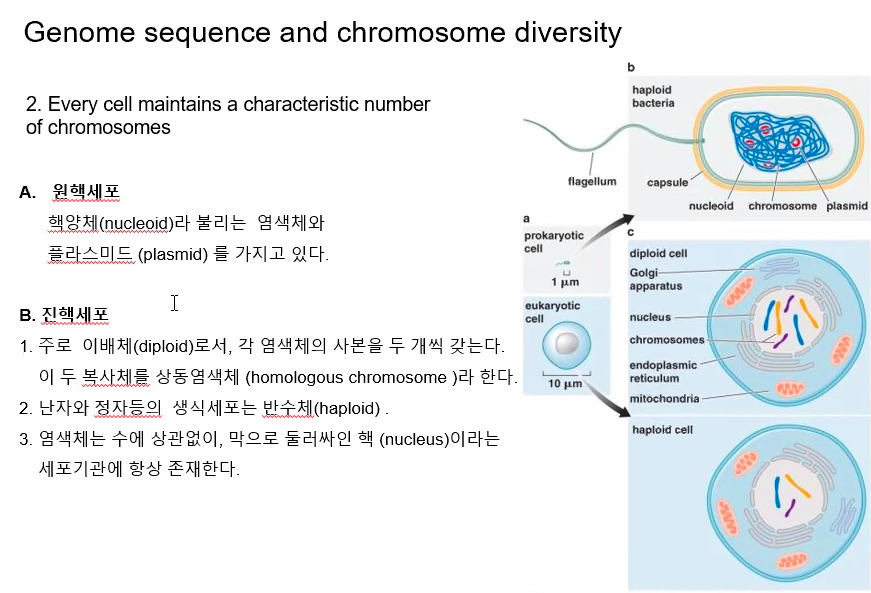
원핵세포는 핵양체(nucleoid)라는 염색체를 가지고 있다.
플라스미드를 가지고 있기도 하다.
핵양체 : 핵과 유사한 물체
진핵세포는 이배체(diploid)이며 주어진 염색체의 두 복사체를 상동체(homolog) 라고 한다.
진핵세포는 내부공간을 나누어 놨다.
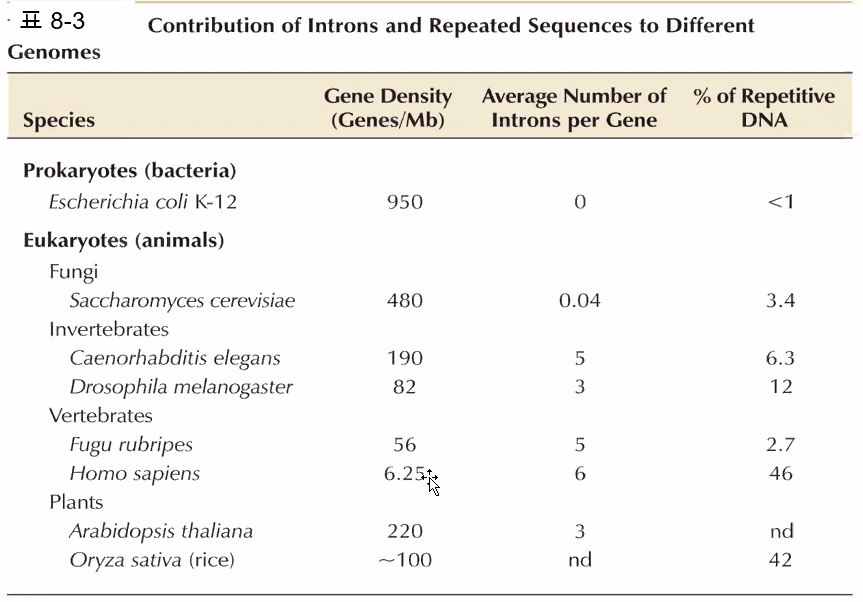
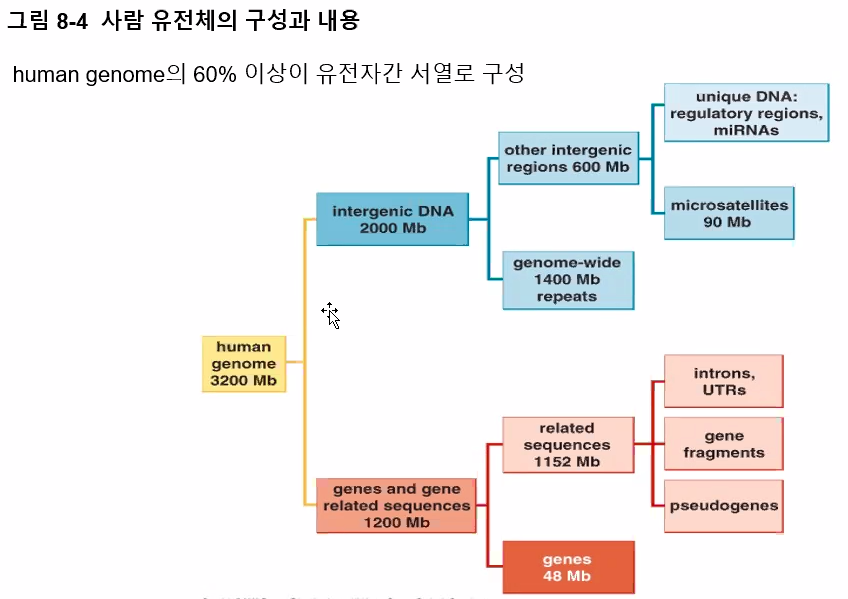
유전체의 크기는 생물의 복잡성과 연관되어 있다.

1. 비암화서열은 바로 뒤의 암호화부위의 전사조절부위로 작용한다.
Ex) 프로모터
2. 유전자 하나가 차지하는 부위가 훨씬 넓다.
생물체의 복잡성과 유전자의 밀도 사이는 역관계가 성립한다.
대장균의 유전체는 대부분이 유전자로 구성되어 있다.
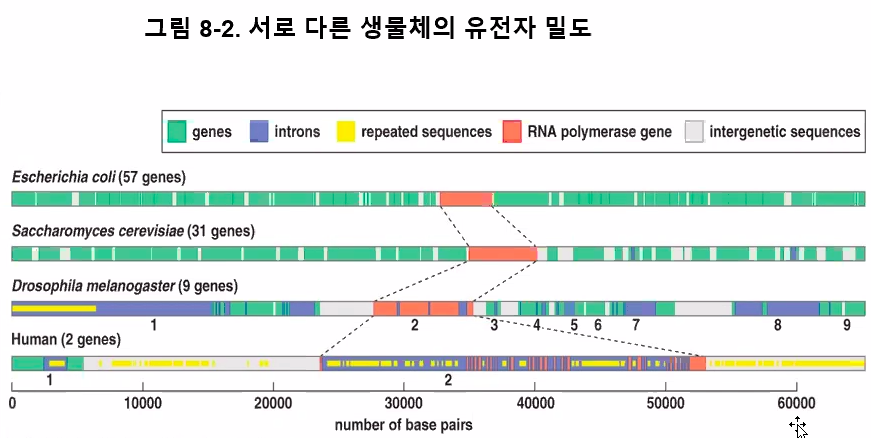
복잡한 생명체는 낮은 유전자 밀도를 가진다.
고등한 생물이 될수록 유전자의 밀도가 낮아진다.
인트론의 부위가 많아지기 때문이다.
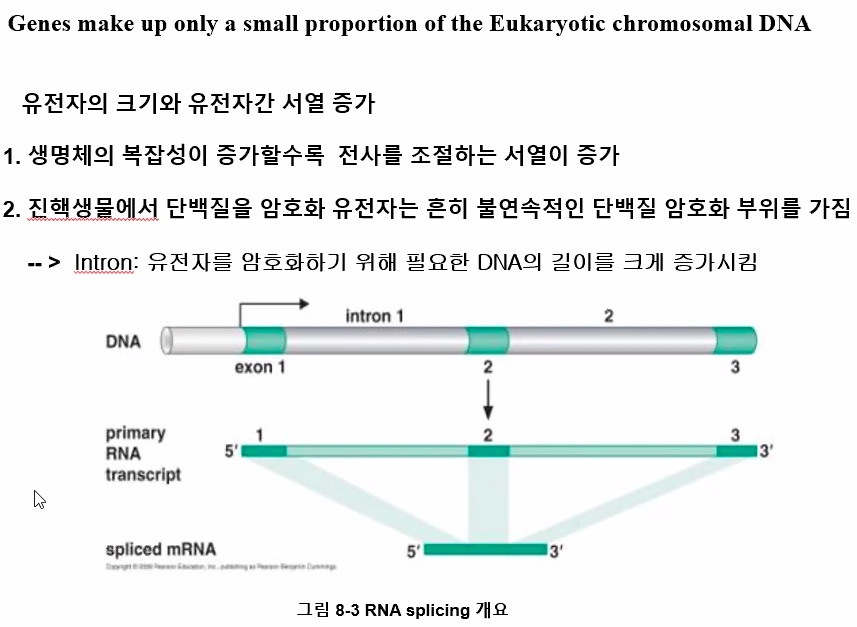
유전자는 진핵생물의 염색체 DNA에서 단지 작은 부분만을 차지한다.
인트론의 존재는 유전자를 암호화하기 위해 필요한 DNA의 길이를 크게 증가시킨다.
사람의 유전자간 서열은 대부분이 반복 DNA로 구성되어 있다.
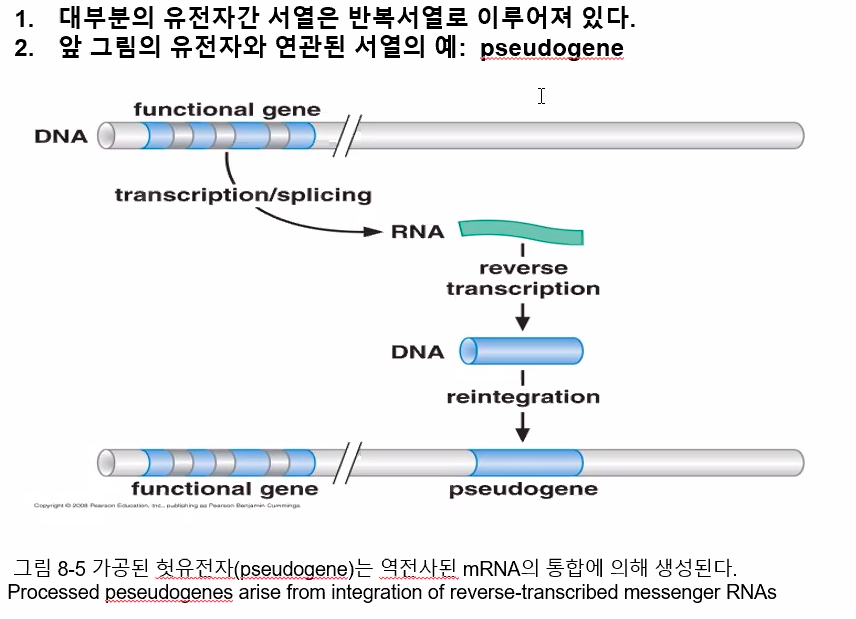
역전사효소로 만들어진 역전사된 DNA는 번역이 되지 않는다.
인트론이 없으니 프로모터도 없기 때문이다.
이것을 pseudogene 이라고 한다.
진핵생물의 염색체가 세포분열 동안에 유지되기 위해서는 동원체, 말단소체 그리고 복제개시점을 필요로 한다.
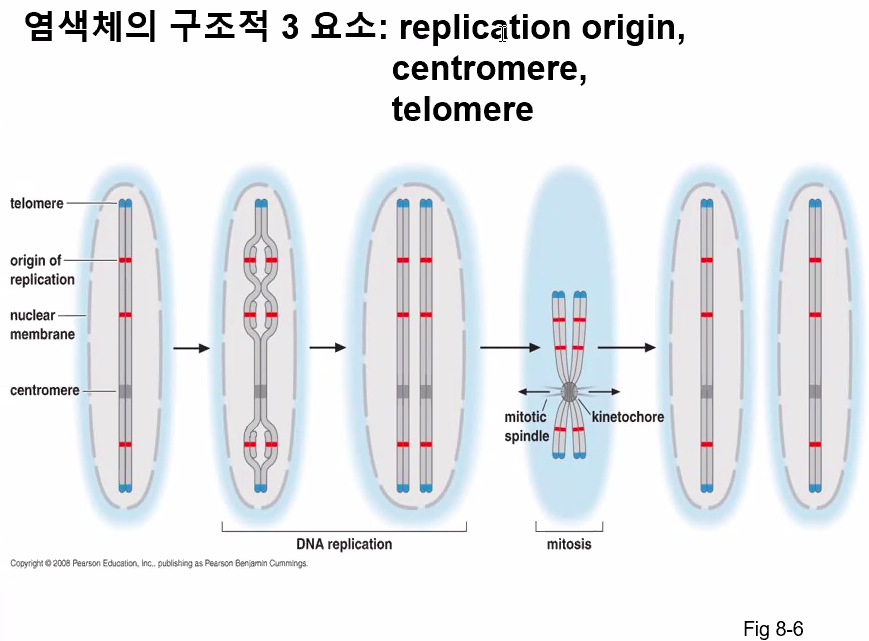
1. Replication Origin (복제 원점) : DNA 복제에 관여하는 요소들이 복제를 시작하기위해 결합하는 장소
2. Centromere (동원체) : DNA 복제 후에 염색체의 정확한 분리에 필요하다.
3. Telomere (텔로미어) : 선형 염색체의 두 말단에 위치한다.
염색체의 본래 말단부위와 세포 내에서 염색체 파손이나 기타 DNA 파괴에 의해 생긴 말단 부분을 구분해준다.
텔로미어는 특수한 복제개시점으로 작용하여 염색체 말단의 복제를 가능하게 해준다.

복제원점에는 유전자가 없다.
동원체 : 복제 후 염색체가 딸세포로 분리될 때 미세소관으로 이루어진 방추사가 결합하는 자리
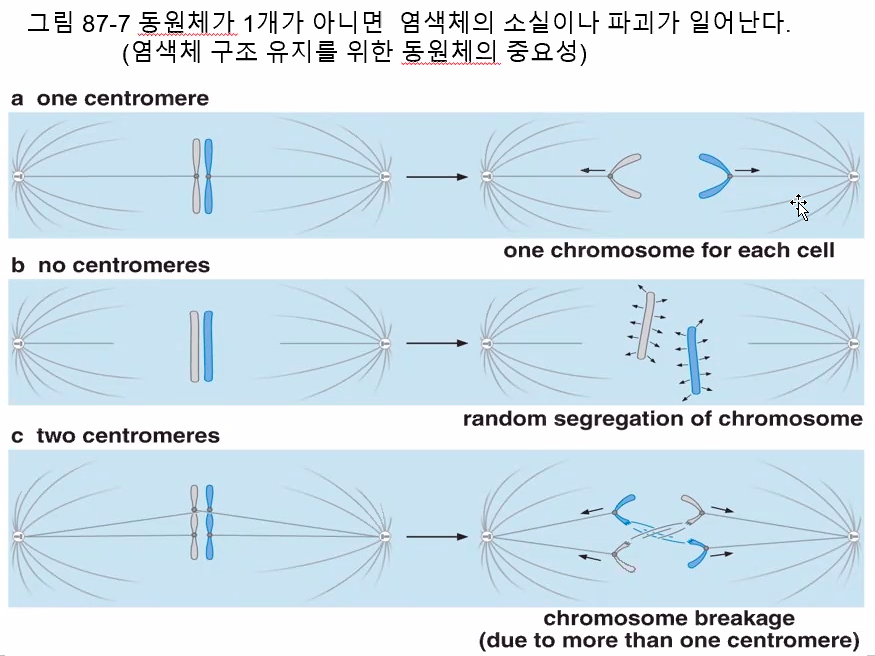

진핵생물 염색체의 복제와 분리는 세포주기의 구분된 단계에서 일어난다.
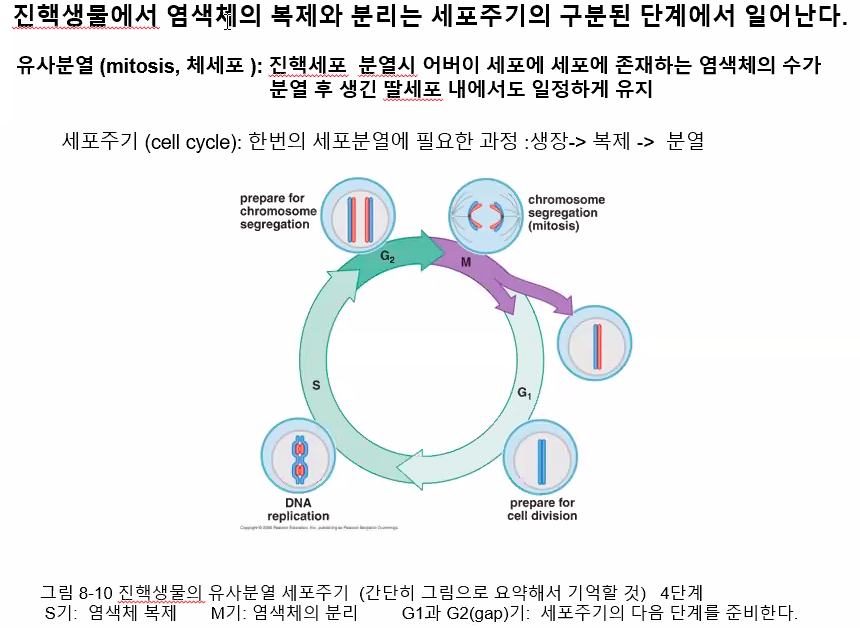
진핵세포의 분열에 따라 염색체의 구조도 변화한다.
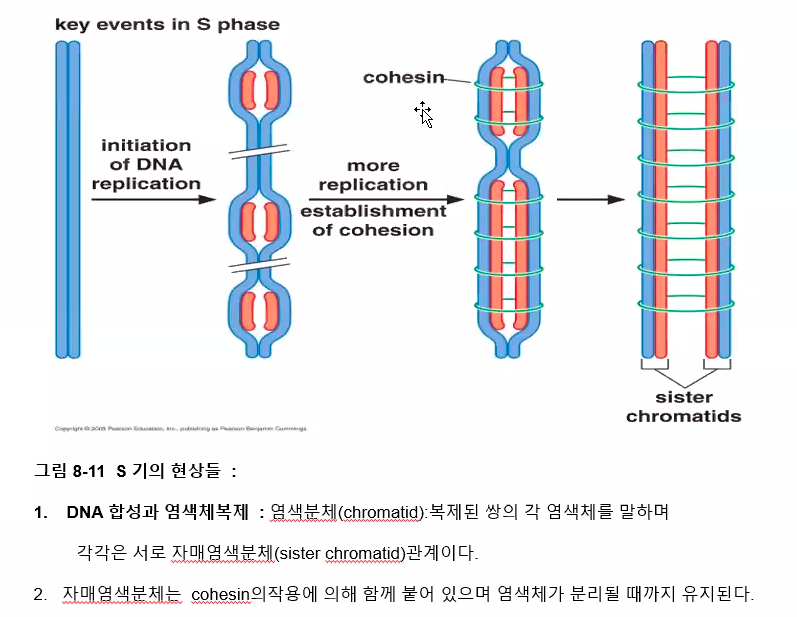
체세포분열이나 감수분열을 거치는 동안 염색체는 압축된 형태로 존재한다.
이러한 압축된 형태를 형성하는 과정을 염색체 응축(chromosome condensation) 이라고 한다.
자매염색분체의 결합과 염색체의 응축에는 Condensin이 관여한다.
Cohesin이라는 것이 염색분체를 묶고 있다.
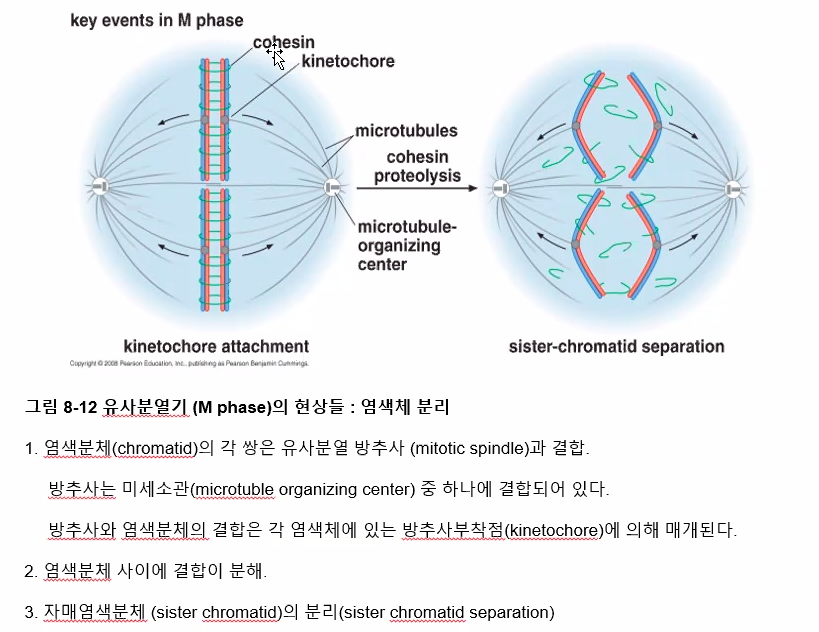
자세하게는 x
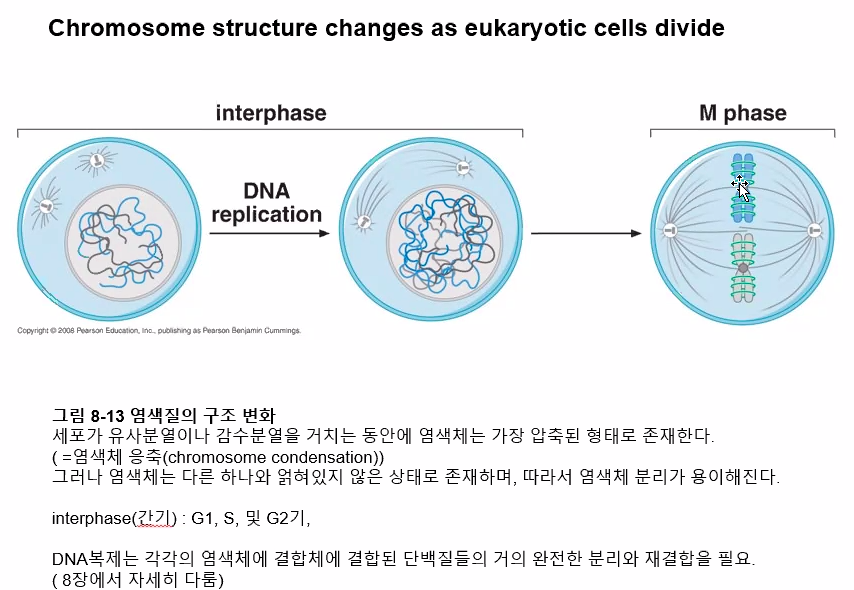
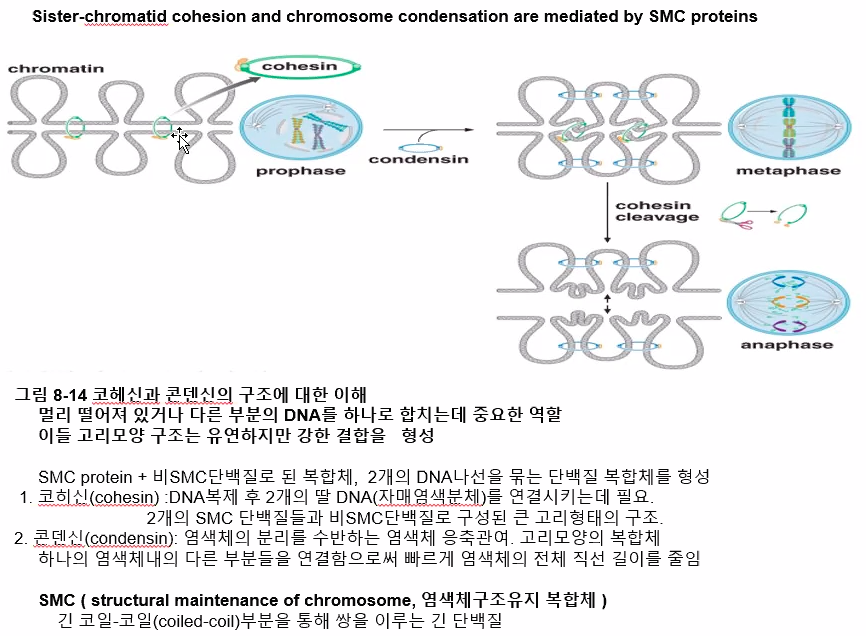
코헤신과 콘덴신
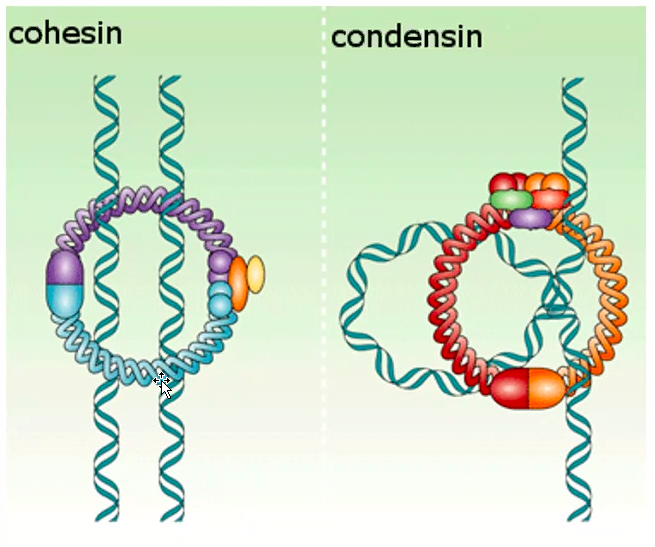
코헤신 : 별개의 염색 분체를 묶음
콘덴신 : 하나의 분자를 고도로 응축시킴
DNA의 입체구조가 어떻게 하위구조에서 상위구조로 넘어가는지 이해해야한다.
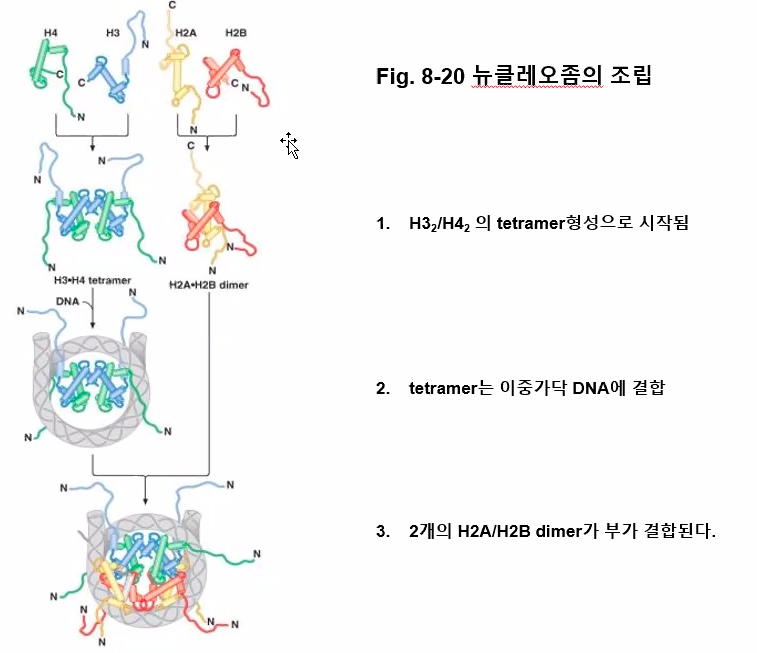
뉴클레오솜은 염색체의 기본 구성 단위이다.
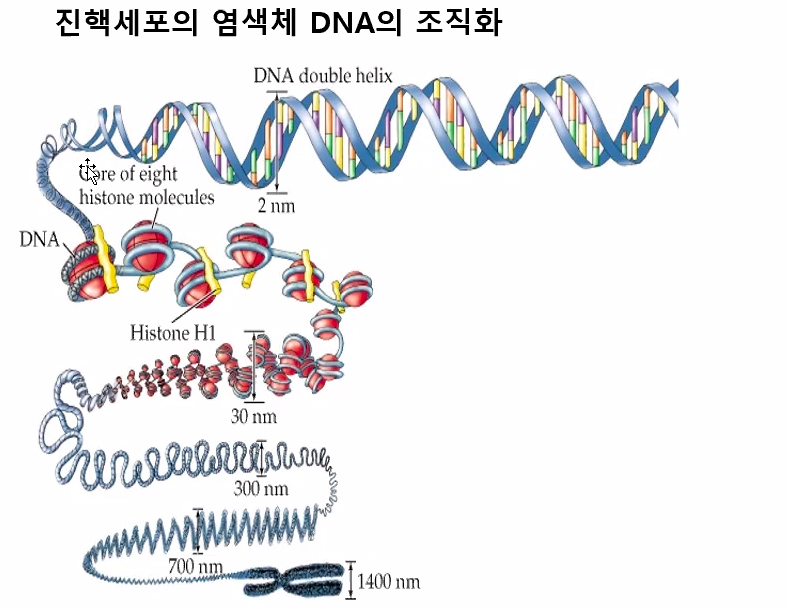
300nm의 루프들이 응축되게 해주는게 콘덴신이다.
1400nm의 접착제역할을 하는 것이 코헤신이다.
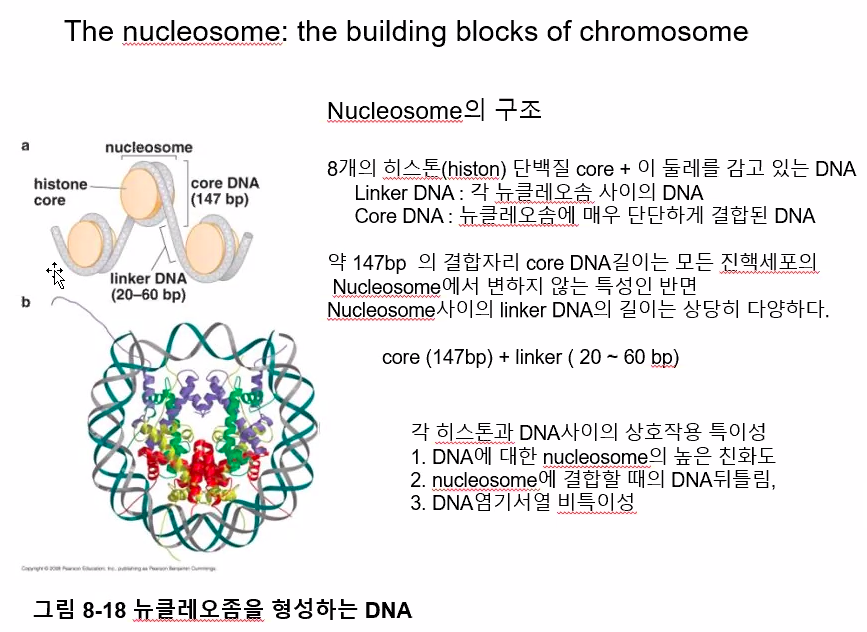
뉴클레오솜 사이의 linker DNA의 길이는 상당히 다양하다.
core DNA길이는 모든 진핵세포에서 147 염기쌍 정도로 DNA 길이가 일정하다.
DNA가 단백질에 대해 친화도를 가지고 있어야 한다.
히스톤은 작고 양전하를 띤 단백질이다.
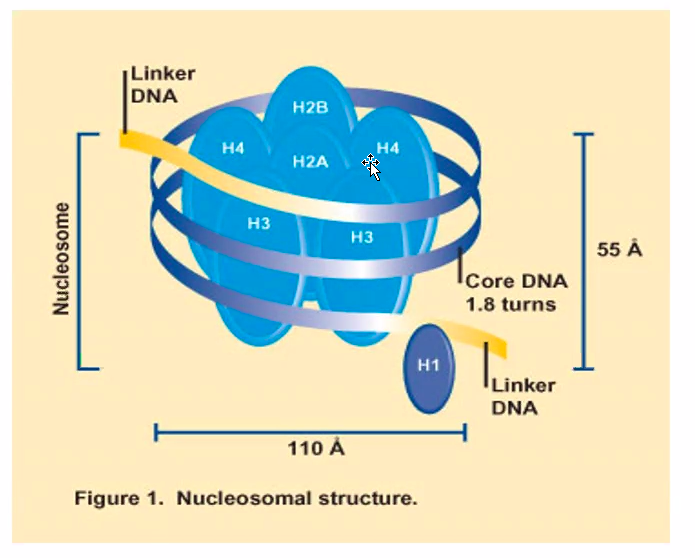
뉴클레오좀은 히스톤 8개가 있다.
H1, H2A, H2B, H3, H4(5종류)
3개는 그림에 안 나옴

뉴클레오솜 하나의 단위 DNA 크기
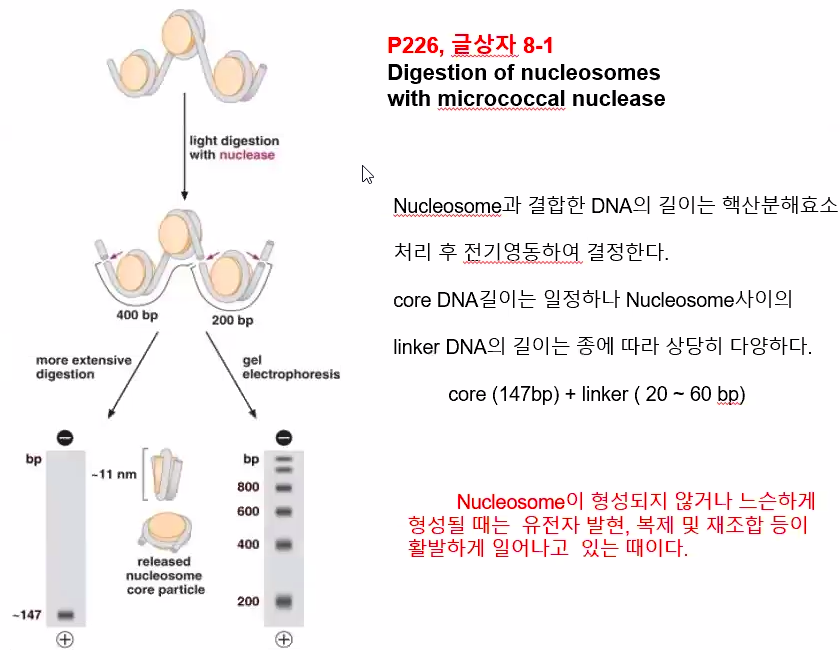
글상자 잘 읽어보기
Core 부분만 안잘린다
히스톤을 제거하면 147bp짜리 DNA 조각을 얻을 수 있다.
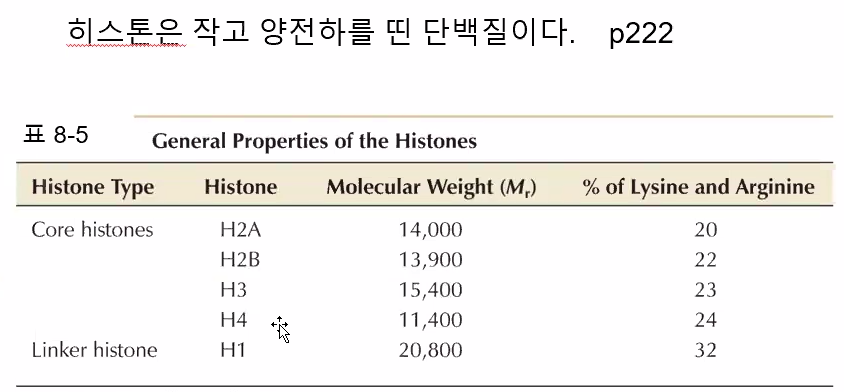
핵 안에서 (+) 를 띠는
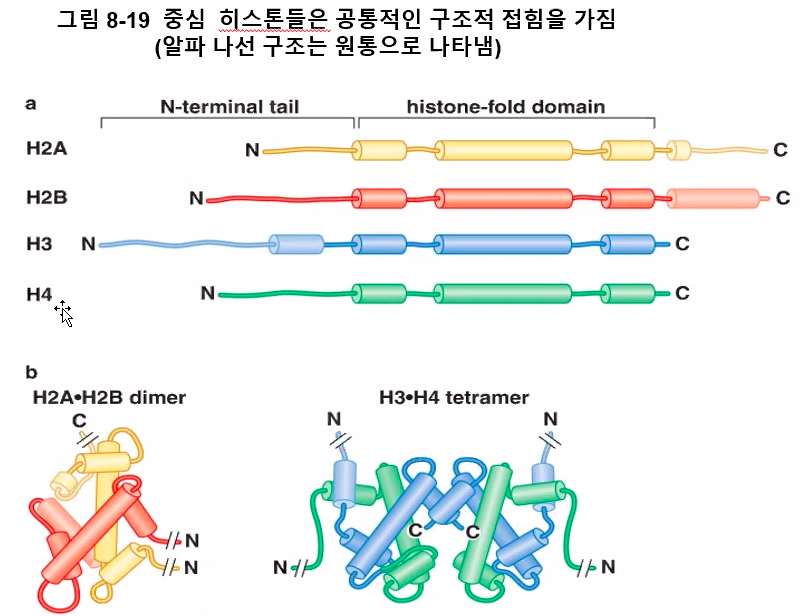
N 꼬리 부분은 안으로 있지 않고 빠져나와 있다.
중요한 역할 한다.

1, 2, 3 중요
N말단이 잘려도 강한 결합이 가능하다.
뉴클레오솜의 구조
뉴클레오솜은 히스톤(Histone) 단백질과 DNA 이중 사슬이 결합하여 규칙적인 구조를 이루는 것으로, 염색체를 이루는 기본 단위가 된다.
DNA는 히스톤 단백질을 1.65바퀴 감는 구조를 하고 있으며, 이것은 위상학적으로 토로이드형(Toroidal) 음성 초꼬임 형태다.
구조는 대략 이러하다.
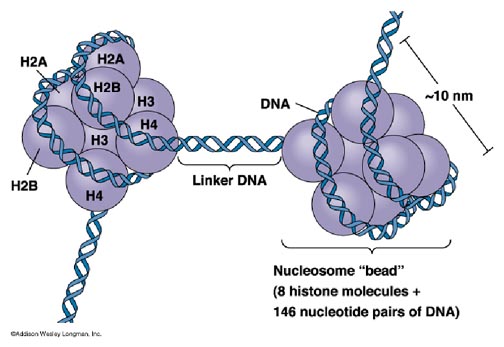
보라색으로 된 구슬같은 것이 히스톤 단백질로, 8개의 서브도메인을 갖는 팔량체(Octamer)다.
히스톤에 감겨지는 DNA의 길이는 ~146bp인데, 이 DNA를 중심 DNA(Core DNA)라 부르며, 뉴클레오솜을 서로 연결하는 노출된 DNA 부위는 연결 DNA(Linker DNA)라 부른다. 연결 DNA의 길이는 평균적으로 대략 20~60bp 정도다.
(이 길이는 전기영동을 통해 밝혀냈다)
중심 DNA의 길이는 대부분의 진핵세포에서 거의 일정하다. 그러나 연결 DNA의 경우 종에 따라 굉장히 다양하다.
효모의 경우 13~18bp, 노랑초파리는 약 33bp, 사람은 38~53bp 정도다.
뉴클레오솜 구조는 DNA의 보존과 보관에 있어 매우 중요하지만, 모든 DNA가 뉴클레오솜을 이루고 있지는 않다.
대표적으로, DNA 전사, 복제, 재조합 등이 일어나는 부분에서는 뉴클레오솜 구조가 풀어져야 한다.
그러나 이런 경우 여러 비히스톤 DNA 결합 단백질(샤페론, Charperon)과 결합하여 DNA의 구조를 안정시키는 경우가 많다. 특히 단일 사슬 DNA의 경우 SSB(Single strand binding protein) 단백질의 역할이 아주 중요하다.
바로 전 포스트에서 DNA는 음전하를 띄기 때문에 응축시키기가 상당히 어렵다는 말을 했다.
그러므로 당연히 정전기적 척력을 상쇄시키기 위해 히스톤 단백질은 양전하를 띄고 있어야 할 것이다.
(리신, 아르기닌 등의 양전하를 띈 아미노산이 외부에 위치하고 있다)
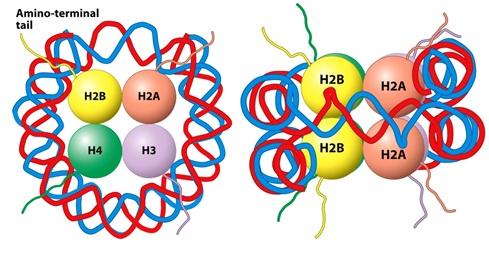
히스톤 단백질에는 5 종류의 서브도메인이 있는데, 각각 H1, H2A, H2B, H3, H4다.
이 중, H1을 제외한 H2~H4가 저기 위 모식도에 나타난 중심 히스톤(Core histone)을 형성한다.
그럼 H1은요? H1 히스톤은 연결 DNA에 결합하여 염색질이 좀 더 컴팩트하게 응축되도록 한다. 뒤에 설명하게 될 것이다. 어쨌거나, 그래서 H1 히스톤은 따로 연결 히스톤(Linker histone)이라 부르기도 한다.
중심 히스톤을 형성하고 있는 8량체는 완전히 면대칭 구조를 하고 있으며, DNA가 오른쪽 위쪽에서 내려와 히스톤을 한바퀴 감고 다시 왼쪽 위로 빠져나간다고 했을때, 대략 다음과 같다.
↑ ↓
이 중심 히스톤 단백질들은 먼저 이량체(Dimer), 또는 사량체(Tetramer)를 이루는데 먼저 H3, H4가 합쳐져서 H3·H4 이형이량체(Heterodimer)를 이루고, H3·H4 이형이량체 둘이 모여 H3·H4 사량체를 형성한다.
한편, H2A, H2B 또한 H2A·H2B 이형이량체를 형성하는데 이 분자들은 사량체는 형성하지 않는다. 대신 DNA와 결합한 H3·H4 사량체에 추가적으로 H2A·H2B 이량체가 2분자 결합하여 팔량체를 형성한다.
DNA와 히스톤 단백질 사이의 결합은 순서대로 다음과 같다.
H3 - H2A·H2B - H4·H3 - H2A·H2B - H3
13bp 30bp 60bp 30bp 13bp
물론 이게 정말 크게 중요한건 아니다(...) 그러나 여기에서 보면, 공통적으로 들어오고 나가는 부분이 H3 서브도메인인 것을 알 수 있다. 여기에는 뭔가 중요한 의미가 있을 것 같지만, 이 포스트에서는 다루지 않는다.
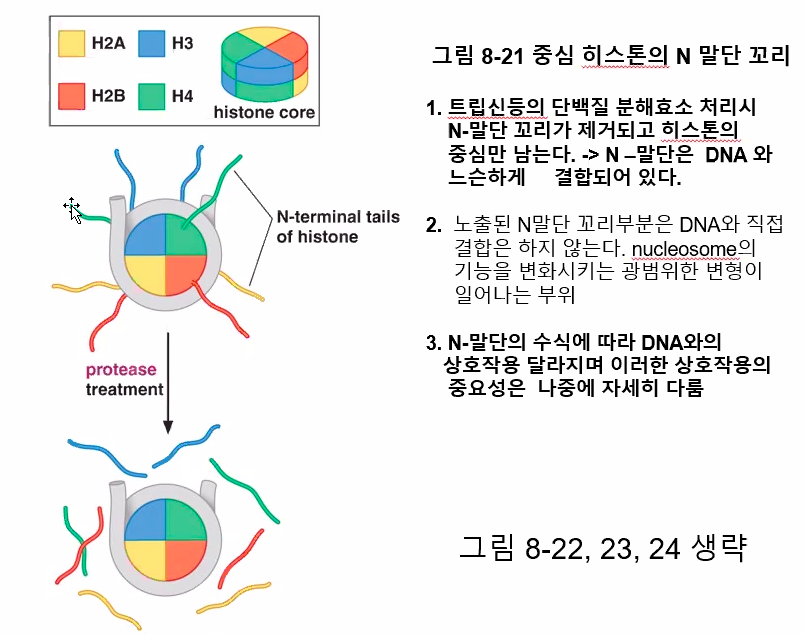
그리고 윗 모식도에서 아미노산 말단 사슬(Amino-terminal tails)들이 삐죽삐죽 튀어나와 있는 것을 볼 수 있다. 이 사슬들은 히스톤 단백질의 구조에는 별다른 영향을 끼치지 않는다.
물론, 저 사슬의 구조상 DNA가 히스톤 단백질을 저러한 형태로 감싸도록 유도하는 역할은 할 수 있지만 일단 뉴클레오솜의 형성이 끝나면 효소로 저 사슬들을 모두 잘라버려도 별 문제가 생기지 않는다.
하지만 저 아미노산 사슬들은 메틸화, 아세틸화, 인산화 등의 다양한 변형(Modification)이 일어나는 부위로, 뉴클레오솜의 특수한 기능을 수행하는 역할을 한다. 물론 이것 역시 이 포스트에서는 자세히 다루지 않는다.
그러면, 어쨌거나 히스톤 단백질은 DNA와 어떻게 결합을 하는가?
히스톤 단백질과 DNA는 수많은(~40) 수소결합에 의해 결합하게 된다. 대부분의 수소결합은 히스톤 단백질과 DNA의 뼈대를 이루는 인산기 사이에서 형성되는데 이 수소결합의 힘에 의해 DNA가 히스톤을 감는 형태의 결합이 이루어진다.
재미있는 것은, 수소결합의 대부분은 DNA의 좁은 홈(Minor grooves)을 형성하는 인산기에서 이루어진다는 점이다. 이것은 염기 서열과는 무관한 결합. 다시 말하자면 염기서열 비특이적인 결합이라는 뜻이다.
(DNA 구조 포스트에서 DNA의 넓은 홈-Major grooves-에는 염기 서열에 대한 정보가 풍부하다고 했던 것 기억하나?)
생각해보면 당연하다. 히스톤 단백질이 염기서열에 특이적으로 결합한다면, 히스톤 단백질들 굉장히 다양한 형태를 띄어야 할 것이고, 이것은 매우 비효율적이다.
또한, 나중에 설명하겠지만 뉴클레오솜에 작용하는 여러 효소는 뉴클레오솜을 마구 변형시키는데, 이 역시도 DNA와 히스톤 단백질이 염기서열과는 무관한 결합을 해야 원활히 이루어질 수 있다.
마지막으로, 맨 위에서 잠시 언급했던 내용으로 뉴클레오솜을 형성하는 것은, DNA에 있어 일종의 음성 초꼬임을 갖는 것을 뜻한다.
무슨 뜻인가? 완전히 풀린 상태(Fully relaxed form)의 cccDNA에 히스톤 단백질을 첨가하고 뉴클레오솜의 조립을 해보자.
그러면 뉴클레오솜이 형성되어 음성 초꼬임이 일어남에 따라(=부분적으로 Wr이 감소함에 따라), 뉴클레오솜의 조립이 일어나지 않은 DNA 고리 부분에서는 양성 초꼬임이 일어나게 될 것이다(=부분적으로 Wr이 증가하게 된다)
그러면 이번엔 위상이성질체 효소를 추가하여 양성 초꼬임이 일어난 부분을 풀어보자.
그리고 뉴클레오솜을 분해하여 히스톤을 제거하면, 음성 초꼬임이 일어나 부분적으로 DNA 사슬의 분리가 일어나거나, 서로 얽힌 형태의 초꼬임을 형성하게 될 것이다.
이것은 무엇을 시사하는가?
진핵세포의 DNA는 선형 DNA지만, 뉴클레오솜의 조립을 통해 음성 초꼬임의 형태를 취한다. 이것은 DNA의 보관과 보존에도 유용하지만, DNA의 전사나 복제에 있어서도 유용하다는 뜻이다.
(전사하거나 복제하기 위해선 DNA가 단일 사슬로 풀려야 하는데 음성 초꼬임에선 DNA가 풀리려 하는 경향이 있다)
다음 내용에서는 이들 뉴클레오솜이 더욱 응축하여 더 높은 차원의 구조를 형성하는 것을 살펴보도록 하자.
고단계의 염색질 구조
여기에선, 히스톤(Histone) 단백질에 의해 1차적으로 응축된 염색질(Chromatin)이 어떻게 더 높은 수준으로 응축되는가에 대해 살펴본다.
초기에, 현미경을 이용한 연구에서 학자들은 염색체를 염색이 잘 되는 부분을 이질염색질(Heterochromatin)이라 부르고, 염색이 잘 되지 않는 부분을 진정염색질(Euchromatin)이라 불렀다.
뒤이은 연구에서 이질염색질 부분은 염색은 잘 되지만 DNA의 전사나 발현은 거의 되지 않는 부분이며, 오히려 염색이 잘 되지 않는 진정염색질 부분에서 DNA의 발현이 활발히 일어남을 알게 되었다.
어째서인가? 그야, 잘 생각해보면 이질염색질 부분은 DNA가 좀 더 응축된 상태로 존재하기 때문에 염색을 하였을 경우 우리 눈에 더 잘 띄었을 것이다.
붉은 실을 이리저리 풀어놓은 것과 실패에 감아놓은 것을 생각하면 된다. 더 응축되었으니 당연히 DNA 전사 효소들이 DNA에 쉽게 접근하지 못하며, DNA의 발현은 제한될 수 밖에 없다.
반대로, 진정염색질 부분은 DNA가 풀린 구조로, 전사와 발현이 좀 더 활발히 이루어질 수 있다.
보통 히스톤 단백질 중 H2A, H2B, H3, H4와 DNA가 결합하여 뉴클레오솜을 형성한 상태의 염색질을 10nm 섬유(10nm fibers)라 부른다.
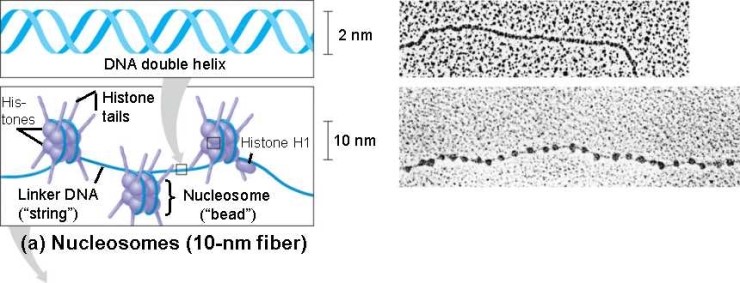
그러나 이것 만으로는 좀 더 높은 수준의 구조를 형성하기 힘들다. 그래서 여기에 추가적으로 결합하는 것이 히스톤 단백질 중 H1 단백질이다.
H1 단백질은 위의 내용에서 연결 DNA(Linker DNA)에 결합하기 때문에 연결 히스톤(Linker histone)이라 부른다고 했었다.
그렇다면 이 H1은 어떤 역할을 하는가?
우선, H1은 뉴클레오솜과 연결 DNA의 사이 부분에 결합하여 DNA를 부분적으로 보호하는 역할을 한다. 핵산분해효소(DNAse)를 처리하였을 때, 20 bp정도의 DNA가 보호됨이 관찰되었다.
그러나 이 역할보다도 중요한 것은, H1이 결합함에 따라 뉴클레오솜들이 서로 더 응축하여 한 단계 높은 수준의 구조인 30nm 섬유(30nm fiber)를 형성한다는 것이다.
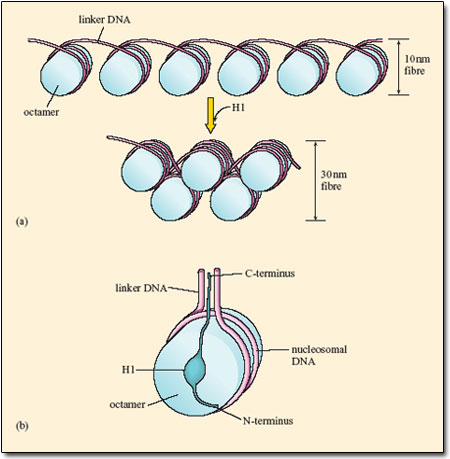
H1은 서로 느슨하게 연결 DNA를 통해 연결되어있던 뉴클레오솜들을 강하게 묶어주는 역할을 한다. 정확하게는, DNA가 들어오고 나가는 각도를 줄이는 것.
DNA의 입출각은 염농도나 pH, 다른 단백질의 유무 등에 따라 다양하다. 그러나 생리적 조건에서 10nm 섬유에 H1을 첨가하게 되면 자연스럽게 위 사진과 같은 30nm 섬유를 형성하게 된다.
30nm 섬유의 종류에는 두 종류의 모델이 있다. 하나는 솔레노이드 모델(Solenoid model)이며, 다른 하나는 지그재그 모델(Zig-zag model)이다.
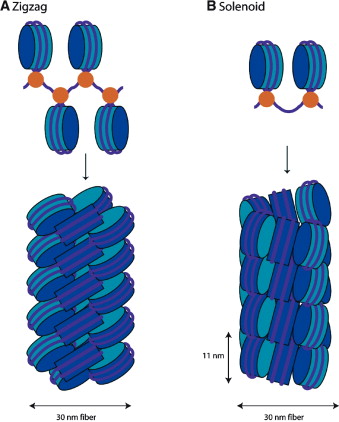
현재까지 여러 연구에 의해 이 두 모델을 나름대로 지지하는 근거가 있지만, 아직까지 이 둘 중 정확히 어떤 형태를 취하고 있는지는 모른다. 즉, 30nm 섬유를 이루긴 하지만 정확히 어떤 모양인지는 모른다는 것.
아직까지도 모델일 뿐이므로, 그냥 이런 것이 지지되고 있다고 받아들이고 넘어가면 될 것이다. 새로운 증거들이 나오면 얼마든지 뒤집힐 수도 있기 때문. 그러니까 저 모델의 특징이나 특성에 대해서 굳이 매달릴 필요는 없다고 본다.
그리고 위에서 뉴클레오솜을 연결하는 연결 DNA의 길이는 종마다 다르다고 했었다. 그러면 당연히 30nm 섬유의 형태로 종마다 약간씩 다르게 나타날 것이다.
만약 연결 DNA의 길이가 상당히 긴 종이라면, 위의 모델 외에 다른 형태의 30nm 섬유를 형성할 수도 있다.
자, 그렇다면 중심 히스톤(Core histone)에서 삐죽삐죽 튀어나온 N-말단 꼬리는 30nm 섬유의 형성에 필요할까?
10nm 섬유에선 일단 뉴클레오솜만 형성하면 잘라버려도 구조가 무너지지 않는다고 했었다.
아미노산 꼬리들은 보통 양전하를 띄고 있다. 그리고 중심 히스톤 단백질의 접힘 구조(Folding structure) 내부는 약간의 음전하를 띈다.
특히, 양전하를 띄는 H4의 아미노산 꼬리와, 이웃한 뉴클레오솜의 음전하를 띄는 H2A 접힘 구조 사이의 정전기적 인력이 30nm 섬유 구조를 지지하는 힘이 된다고 알려져 있다.
이게 대체 무슨 말인가?
H1을 넣으면, 일단 DNA의 입출각이 좁아져 뉴클레오솜끼리 서로 맞붙게 된다. 그러나 위의 두 구조처럼 규칙적인 형태를 띄기 위해선 그 구조를 지지하는 힘이 필요하다는 뜻이다.
이 구조를 지지하는 것이 중심 히스톤의 N-말단 꼬리(양전하)와 중심 히스톤 내부 구조(음전하) 끼리의 정전기적 인력이다.
어쨌거나 H1의 첨가를 통해 30nm 섬유를 형성하게 되면, 이제 슬슬 DNA에 관련되는 여러 효소가 제대로 일을 못 하기 시작한다. 딱 봐도 DNA가 서로 엄청나게 꼬여있는데 저 상태에서 뭔갈 하는 것은 무리일 것이다.
만약에 DNA의 전사를 위해 30nm 섬유 구조를 풀고 싶다면 어떻게 하면 될까? H1을 제거? 쉽지 않을 것이다.
해답은 중심 히스톤의 N-말단 꼬리를 변화시키는 것에 있다.
아미노산 말단을 아세틸화하거나, 메틸화하거나, 인산화하는 것인데, 각각의 단계에서 어떤 변화가 일어나는가?
우선, 아세틸화의 경우 N-말단 꼬리의 양전하가 상쇄된다. 그 결과 30nm 섬유 구조가 풀려 느슨해지게 된다.
반대로, 메틸화의 경우 N-말단 꼬리의 양전하가 증가하여 30nm 섬유 구조는 더 강력해지고(...) 그 결과 DNA의 불활성화가 일어날 수도 있다.
인산화의 경우는 아세틸화와 마찬가지로 N-말단 꼬리의 양전하가 상쇄되어 전사를 촉진시킬 수 있으나, 대부분의 경우 전사를 촉진하기 위해서는 아세틸화를 많이 하게 된다.
이 부분은 나~중에 DNA 전사 조절에 대해서 다룰 때 자세히 다룰 예정이다.
여튼, 이 단계에서 DNA의 길이는 약 40배 정도 직선 길이가 압축될 수 있다. 그러나 이 정도로는 충분하지 않다.
좀 더 높은 수준의 응축이 필요하다. 그러나 다행스럽게도(?) 이 구조에 대한 연구는 아직 완전하게 이루어지지 않았다. 구조의 형태는 알게 되었으나 자세한 사실은 아직 모르고 있다. 그러니 안도하도록 하자(...)
그래도 이미 알려진 사실은 짚고 넘어가야 하지 않겠는가.
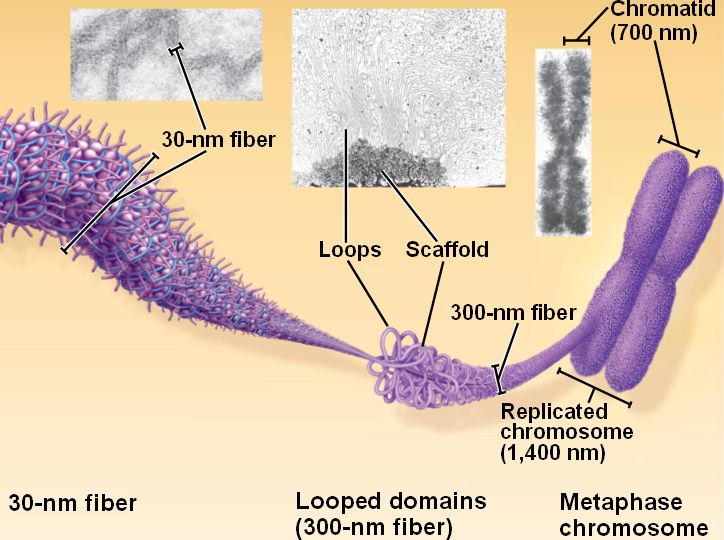
30nm 섬유 다음의 고리형 영역(Looped domains)은, 30nm 섬유가 가운데의 핵골격(Nuclear scaffold)에 의해 고정되어 40~90kb의 고리형 구조를 형성하고 있는 형태다.
여기에서 상당히 재미있는 특징이 나타나는데, 저 고리형 구조를 이루는 섬유는 30nm 섬유로만 이루어진 것이 아니다.
핵골격에 고정된 부분은 30nm 섬유지만 점점 중간쪽으로 가면서 구조가 풀려 10nm 섬유가 되거나, 심지어는 아예 노출된 DNA(Naked DNA)가 나타나 있기도 한다.
이것은 무엇을 뜻하는가?
30nm 섬유의 고리형 구조에서 중간중간 풀린 부분이 진정염색질로써, 전사가 활발히 일어나는 지역이다. 반대로, 핵골격 근처의 강력한 30nm 섬유 밀집부위는 이질염색질이 된다.
그렇다면 어떻게 해서 저런 것들이 조절이 되나요? 글쎄 그것은 모르겠다. 현재로는 핵골격 단백질이 어떤 녀석인지도 확실하게 알아내지 못 했다.
그러나 핵골격에 영향을 주는 단백질은 몇 가지 발견했는데, 그 중 하나가 제 2형 DNA 위상이성질화 효소(DNA topoisomerase Ⅱ)다.
제 2형 효소는 특히 원심분리로 분리한 핵골격과, 추출해낸 염색체(우리가 아는 그 X형태 염색체 맞다) 내부에 상당히 많이 존재한다.
또한, 제 2형 효소가 결합하는 부위를 절단하는 DNA 분해효소를 처리할 경우 나타나는 DNA 조각들과, 분리해낸 염색체에 동일한 효소를 처리했을 경우 나타나는 DNA의 조각 크기가 유사하다.
이 것은 어떤 점을 시사하는가? 제 2형 위상이성질체 효소가 핵골격 내에서 30nm 섬유를 붙잡고 있을 수도 있다는 점을 시사한다.
그리고 코헤신(Cohesin)이나 콘덴신(Condensin)과 같은 SMC(염색체 구조유지, Structure maintenance of chromosome) 단백질들 역시 핵골격에 매우 풍부한 구성요소임이 확인되었다.
이들은 염색질 구조에서 간단히 본 것과 같이 염색체의 응축과 분리에 있어 핵심적인 역할을 담당한다. 따라서, 핵골격의 유지와 DNA와의 상호작용에 있어 어느 정도 기여를 하고 있는 것으로 여겨진다.
다만, 이것들 역시 아직 명확한 역할이 밝혀진 것은 아니다. 여러 근거를 토대로 하여 추측의 단계에 머무르고 있다.
'🧬 Bio > 분자생물학' 카테고리의 다른 글
| 분자생물학 9장 (2) - 복제분기점, Primase, Helicase, DNA polymerase (6) | 2020.10.10 |
|---|---|
| 분자생물학 9장 (1) - DNA 복제 (0) | 2020.10.04 |
| 분자생물학 8장 (2) (0) | 2020.09.25 |
| 분자생물학 4장 - DNA 입체구조 (2) (3) | 2020.09.11 |
| 분자생물학 4장 - DNA 입체구조 (1) (0) | 2020.09.08 |