잘 생각해보면 굳이 번역하는 걸 조절할 이유가 있을까 싶기도 하다. 어차피 필요한 단백질을 만들기 위해 전사의 조절이 일어나니까.
필요해져서 만든 mRNA를 세포질까지 꺼내와 놓고 필요 없다고 꺼버리는 것은 어떻게 생각해보면 낭비라고 생각할 수도 있다.
그러나 mRNA들 마다 번역을 많이 돌려서 많은 단백질을 합성해야 하는 mRNA가 있고, 조금만 만들고 폐기해야 할 mRNA가 있다.
특히 전사 수준에서 조절하게 되면 아무래도 외부의 환경 변화에 대한 반응이 늦기 마련이다.
따라서 기껏 전사해서 보내놨더니 막상 필요없어지는 경우도 있을 것이다. 이런 여러 이유로 번역의 조절이 필요하다.
조절은 따로 촉진되는 기전은 뚜렷하지 않고 주로 억제하는 방향으로 이루어지는데, 많은 경우 합성한 단백질이 억제 인자로써 작용하는 경우가 많다.
단백질을 일정 수준 이상으로 합성하게 되면 음성 되먹임과 유사한 기전을 통해 리보솜의 번역이 억제되는 기작이 가장 일반적이며, DNA에 작용하여 전사 자체를 억제하기도 한다.
지금까지 보면 눈치챘겠지만 전사의 조절에 대해선 상세히 다루지 않았다(...) 아마 나중에 다룰 기회가 있을 것이다.
원핵생물의 번역 조절
원핵생물들은 보통 조절 단백질들이 리보솜 결합 부위(RBS) 근처의 서열에 결합하여 리보솜이 RBS에 결합하지 못 하도록 하여 조절을 억제한다.
이것을 이용한 방법의 예가 리보솜 단백질의 번역 조절인데, 리보솜 단백질은 전사된 rRNA의 양 만큼 합성되어야 낭비되지 않을 것이다.
리보솜 단백질은 RNA 결합 단백질로 rRNA에 결합하여 리보솜을 조립한다.
그러나 재미있게도 리보솜 단백질은 자기 자신을 암호화한 mRNA에도 결합이 가능하며, 일종의 번역 저해인자로 작용한다.
하지만 상대적으로 rRNA에 대한 친화력이 더 높아 rRNA와 결합하지 못 한 잉여 리보솜 단백질이 mRNA에 결합하여 번역을 저해한다.
따라서, 리보솜이 모두 조립되어 번역이 중단되었더라도 인위적으로 rRNA를 더 넣어주게 되면 넣어준 rRNA의 양 만큼 단백질이 추가적으로 번역된다.
이 방법은 리보솜 단백질 뿐만 아니라 아미노아실-tRNA 합성효소를 포함한 여러 단백질에서 일어나는 기전 중 하나다.
그러나 그것 외에도 RNA의 구조적 특징에 의한 조절 기전이 더 존재하는데 이것은 원핵생물들의 mRNA 특징을 잘 생각하면 추론해 낼 수 있다.
원핵생물들의 mRNA는 비슷한 목적을 가진 유전자들이 하나의 mRNA에 연속적으로 나타난다.
이것을 뭐라 한다? 폴리시스트론 mRNA(Polycistronic mRNA)라 한다. 오페론(Operon) 유전자군 역시 이것을 이용하여 전사를 조절하는 방법이지 않은가.
폴리시스트론 mRNA에는 여러 개의 열린 읽기틀(ORF)이 존재하여 여러 개의 유전자를 연속하여 합성해 낼 수 있다.
mRNA는 단일 사슬이다. 그냥 내버려 두면 스스로 접히고 꼬여서 2차 구조를 형성한다.
앞에서 원핵생물은 번역이 시작되기 위해선 리보솜이 RBS에 결합해야 한다는 설명을 한 바 있다.
그러나 mRNA의 5' 말단 쪽 첫 RBS에는 리보솜이 쉽게 결합할 수 있겠지만 중간에 위치한 RBS들은 RNA가 고리나 헤어핀 구조를 형성하여 리보솜이 결합하기 곤란할 것이다.
노출되어 있는 5' 말단 첫번째 RBS에 리보솜이 결합하여 번역을 개시하면, 번역이 진행됨에 따라 자연스럽게 mRNA의 이차 구조가 풀리게 된다.
그러면 하나의 ORF를 번역한 후, 바로 옆에 있는 두번째 RBS에서 다시 번역을 개시할 수 있을 것이다.
따라서, 원핵생물에서의 번역은 하나의 mRNA에 존재하는 모든 유전자들이 하나의 셋트로 취급되어 번역이 진행되는 것이 일반적이다.
다음은? 물론 진핵생물이다. 그렇게 어렵지 않을 것이다.
진핵생물의 번역 조절
예고한 대로, 진핵생물에 있어서는 어떤 기전들이 존재하는가?
앞서 보았던 조절 단백질에 의한 번역의 조절이 일어나기 위해선 어떤 방법이 효과적이겠는가?
원핵생물은 번역 개시가 일어나기 위해 리보솜이 RBS를 인식하고 결합해야 한다.
진핵생물에서는 번역 개시를 위해 5' 말단의 캡에 결합한 신장인자 eIF4E와 그에 결합한 eIF4G, eIF4A를 리보솜이 인식해야 한다.
그렇다면 감이 잡히는가? eIF4E나 eIF4G, eIF4A와 같은 신장인자의 결합을 방해하여 리보솜이 인식하지 못 하게 하면 될 것이다.
4E-BP(eIF4E-binding protein)은 이름 그대로 eIF4E에 결합하는 일련의 단백질군이다. 여러 종류가 있다.
4E-BP는 eIF4G와 경쟁적으로 eIF4E에 결합하는데, 4E-BP의 결합은 번역이 개시되지 못 하게 하는 저해 효과가 나타난다.
그러나 진핵생물은 모노시스트론 mRNA(Monocistronic mRNA)을 주로 갖고 있으며 억제 단백질을 함께 번역하지 않는다.
그러므로 원핵생물과 같은 형식의 음성 되먹임과 비슷한 조절을 바라긴 힘들다.
따라서 진핵생물은 따로 4E-BP 단백질의 활성을 조절하여 번역을 조절하는데, 세포 내부의 인산화효소 중 하나인 mTor라는 효소에 의해 4E-BP를 인산화하여 기능을 정지시킨다.
성장 호르몬과 같은 단백질 합성 촉진인자들은 mTor의 활성을 높여 세포 내 단백질 합성의 수준을 증가시키는데, 특히 분열을 활발히 하는 세포(ex: 암세포)에서는 mTor의 활성이 높다.
(라파마이신-Rapamycin-과 같은 일부 항암제나 항생제는 mTor의 활성을 저해하여 성장을 억제한다)
이것과 유사하지만 mRNA 특이적으로 발현되는 방식도 있는데, 두 종류의 조절 단백질이 필요하다.
Bruno 단백질
노랑초파리의 오스카(Oscar) 단백질을 암호화하는 mRNA의 종결 코돈 하류의 비번역 부위에는 브루노(Bruno)라 불리는 단백질과 결합할 수 있는 서열(BRE, Bruno response element)이 있다.
브루노가 BRE를 인식하여 mRNA에 결합하게 되면 브루노를 인식하고 4E-BP 단백질의 일종인 컵(Cup) 단백질이 브루노에 결합한다.
그러면 브루노와 결합한 컵 단백질은 4E-BP 활성을 갖게 되어 mRNA의 eIF4E와 결합이 가능해지고, 번역이 저해된다.
컵 단백질은 일반적인 다른 4E-BP 단백질보다 그 양이 훨씬 적지만, 특정한 mRNA에 대해 특이적으로 작용하여 효과적으로 번역을 저해할 수 있다.
Nanos 단백질
다른 예로써, 초파리의 나노스(Nanos) 단백질을 암호화하는 mRNA 역시 컵 단백질에 의해 번역이 조절된다.
그리고 또 한 가지 대표적인 진핵생물의 번역 조절 기전으로써 신장인자인 eIF2의 인산화가 있다.
(원핵생물도 신장인자 자체를 조절하는 번역 조절 기전이 있지만 여기선 굳이 언급하지 않았다)
eIF2는 GTP결합 단백질로써, 리보솜으로 개시 tRNA를 끌고 들어오는 신장인자다.
elF2α 인산화효소는 eIF2α 서브유닛을 인산화시키는데, eIF2에 존재하는 eIF2B 서브유닛의 활성을 저해하는 결과를 초래한다.
eIF2B 서브유닛은 eIF2+GDP의 GDP를 GTP로 교환해주는 역할을 담당하는데, 개시 tRNA는 eIF2+GTP 복합체에 의해서 리보솜으로 운반된다.
따라서 eIF2의 인산화는 eIF2-GTP의 수준을 감소시키고, 이것은 번역의 개시를 저해하는 한 가지 방법이 된다.
eIF2α 인산화효소는 세포 내 아미노산이 부족할 경우, 바이러스 감염, 온도의 상승 등 여러 세포 내 조건에 의해 활성화되어 전체 번역 수준을 조절한다.
페리틴 유전자
그리고 원핵생물의 경우와 마찬가지로 mRNA에 조절 단백질이 결합하여 번역의 진행을 방해하기도 하는데, 인체에 있어 체내 철분 농도를 조절하는 단백질인 페리틴(Ferritin) 유전자를 살펴보자.
페리틴은 헤모글로빈과 마찬가지로 철을 함유한 단백질이며, 세포 내부의 철을 저장하는 역할을 한다.
페리틴 mRNA의 번역은 철분 조절 단백질(IRP, Iron regulatory protein)에 의해 조절되는데, 세포 내부의 철분 농도에 민감하게 반응한다.
IRP는 RNA 결합 단백질인데 페리틴 mRNA의 5' 말단에 위치한 철분조절인자(IRE Iron regulatory element)를 인식하여 결합한다.
IRE 부위는 헤어핀 구조를 하고 있는데 IRP가 이 구조에 결합하게 되면 리보솜이 mRNA에 결합하지 못 하여 번역이 저해된다.
그러나 IRP는 철분에 의해 그 활성이 조절되는데, 세포 내부에 철분이 충분할 경우 철분이 IRP에 결합하여 RNA 결합 활성을 억제한다.
그러면 페리틴은 정상적으로 번역이 진행되어 세포 내의 철분을 수거한다.
반대로, 세포 내의 철분이 부족할 경우 IRP는 RNA 결합 활성을 가지므로 페리틴의 발현을 억제하여 세포 내의 철분 농도를 증가시킨다.
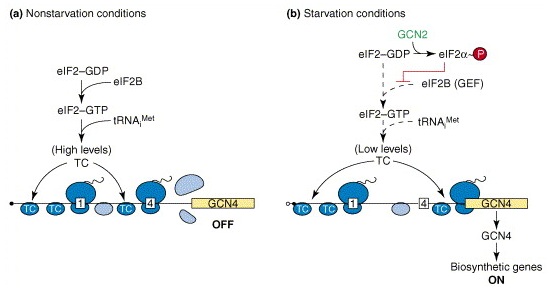
하나만 더 보자. 효모의 Gcn4 유전자 mRNA는 원핵생물과 같은 폴리시스트론 mRNA(polycistronic mRNA)다.
이 유전자는 세포 내의 아미노산 수준에 의해 조절이 되는데, 아미노산이 부족할 경우 번역이 개시되어 전사를 조절하는 단백질이다.
(Gcn4 단백질은 전사 조절 단백질이지만 Gcn4 단백질 자체의 발현은 번역에서 조절된다는 뜻)

이 mRNA에는 Gcn4 유전자를 암호화하는 ORF의 상류에 매우 짧은 4개의 ORF(uORF1~4)를 추가로 갖고 있다.
uORF1~4는 서로 가깝게 모여 있는 짧은 ORF인 반면, 실제 발현이 되는 유전자 부분인 Gcn4 ORF는 uORF들에 비해 상당히 멀리 떨어져 있다.
하지만 그렇다고 ORF들이 원핵생물처럼 각각 RBS(Ribosome-binding site)을 갖는 것은 아니다. 원핵생물과 진핵생물은 전사 개시 방법에 큰 차이가 있다는 것을 기억하자.
이 mRNA는 원핵생물들의 mRNA와 마찬가지로 최상류(5' 말단)에 위치한 ORF부터 번역이 개시되는데, ORF 하나를 번역을 마쳤다고 하여 바로 리보솜이 떨어져 나오지 않는다.
최상류에 있는 uORF1를 번역한 리보솜은 떨어지지 않고 mRNA를 계속 스캐닝하는데, 다음 ORF를 번역하기 위해선 elF2+GTP+개시 tRNA 복합체. 즉, 삼중복합체(TC, Ternary complex)가 리보솜에 결합해야 한다.
재미있는 것은, 리보솜은 TC가 결합하지 않은 빈 상태라도 혼자서 mRNA의 스캐닝을 계속 진행한다는 점이다.
그러나 TC가 결합하지 않을 경우, 리보솜은 개시코돈을 인식하지 못 하므로 TC가 언제 결합하느냐에 따라 두 번째로 번역하는 ORF의 종류가 달라진다.
아미노산이 세포 내에 풍부하게 존재할 경우, TC 역시 높은 수준으로 존재하여 uORF1의 번역이 종결된 후 빈 리보솜에 빠르게 TC가 재결합할 수 있다.
그러면 리보솜은 곧바로 근처에 있는 다른 uORF2~4 중 하나를 번역할 수 있다.
두 번째 ORF까지 번역한 리보솜은 완전히 번역을 종결하고 떨어져 나오게 되어, 그 결과 Gcn4 ORF는 번역이 되지 않는다.
그러나 아미노산이 세포 내에 부족한 경우, Gcn2 단백질이 eIF2α 인산화효소를 활성화시킨다.
그러면 eIF2가 인산화되어 세포 내의 elF2-GTP 복합체의 수준이 떨어지게 되고, TC의 수준 역시 낮아진다.
TC의 수준이 낮아지게 되면, uORF1의 번역을 종결한 빈 리보솜에 TC가 결합하는 속도가 느려지게 된다.
그러면 빈 리보솜은 uORF 부위를 모두 스캐닝하며 지나칠 때까지도 TC가 결합하지 않아 번역을 하지 못 하다가 Gcn4 ORF 앞에서야 TC가 결합할 수도 있을 것이다.
그렇게 되면 Gcn4 유전자가 발현하여 Gcn4 단백질을 합성할 수 있게 된다.
위에서 eIF2α 인산화효소의 활성이 있을 경우 번역이 저해된다고 했었으나, 이것은 그 응용(...)이라고 볼 수 있을 것이다.
물론 큰 그림에서 보자면 아미노산이 부족하게 되면 번역의 수준 자체가 전체적으로 억제된다고 볼 수 있을 것이다.
즉, 아미노산이 풍부하다면 10개의 mRNA가 모두 번역이 진행되어도 Gcn4 단백질은 하나도 중합되지 않는다면,
아미노산이 고갈되었을 때는 10개의 mRNA 중 제대로 번역이 진행된 것은 겨우 3~4개 정도지만 그 결과 Gcn4 단백질이 발현된다는 뜻이다.
그런데 uORF1~4에서 합성되는 폴리펩티드들은 어디에 쓰나요? 글쎄, 그것까진 잘 모르겠다.
'🧬 Bio > 분자생물학' 카테고리의 다른 글
| 분자생물학 (18) - Transcriptional regulation in prokaryotes 2 (0) | 2020.12.06 |
|---|---|
| 분자생물학 (18) - Transcriptional regulation in prokaryotes (0) | 2020.12.05 |
| 분자생물학 (16) - Genetic code (0) | 2020.12.04 |
| 분자생물학 (15) - Translation 2 (0) | 2020.12.04 |
| 분자생물학 (15) - Translation (1) | 2020.12.03 |