
인간의 만들 수 있는 유전자는 20000여개이다. 그러나 인간이 가질 수 있는 서로다른 단백질의 개수는 10만여개가 넘는다.
Alternative splicing : 주어진 숫자의 유전자를 가지고 훨씬 더 많은 단백질을 만드는 것



복잡한 생물로 갈수록 인트론이 늘어난다.

인트론을 제거할 때 덜 제거하거나 더 제거하면 심각한 돌연변이가 만들어진다. 따라서 굉장히 정확하게 만들어야 한다.



Splicing 기구


















Trans-splicing



Minor spliceosome



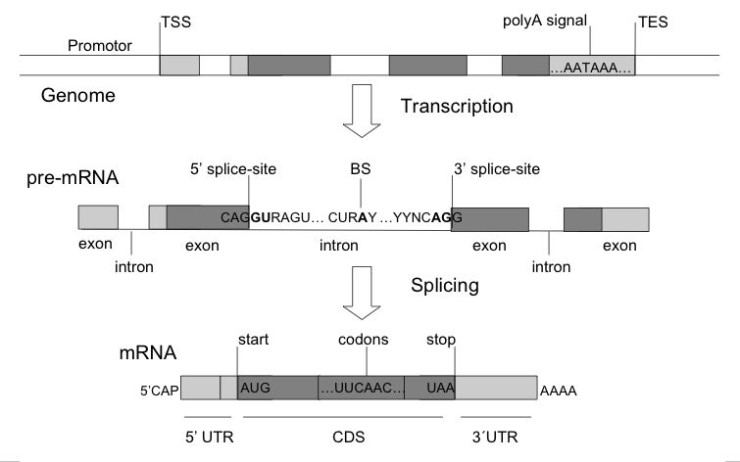
RNA 이어맞추기는 다들 알다시피 중합된 pre-RNA에서 인트론(Intron)을 제거하고 엑손(Exon)만 연결하는 것을 뜻한다.
진핵세포에서만 나타나는 현상으로, 일반적으로 고등한 생물로 갈수록 유전자 당 인트론의 수가 증가하는 경향이 나타난다.
인트론은 보통 엑손보다 훨씬 길이가 길며, 포유류의 경우 실제 엑손은 전체 유전자의 약 10% 이내밖에 되지 않는다.
RNA 이어맞추기
대체 왜 인트론이 있는가? 인트론은 우선 그 긴 길이로 인해 엑손이 손상당할 확률을 확률적으로 줄여준다.
또한, RNA 이어맞추기를 이용하여 엑손들을 선택적으로 이어맞추는 것으로 하나의 유전자에서 다양한 단백질을 발현할 수 있다.
이것은 대체 이어맞추기(Alternative splicing)이라 부르는 과정으로, 미리 말해두지만 굉장히 매우 복잡한 과정이다.
대체 이어맞추기에 대해 간단히 예를 들어 설명하자면 이렇다.
만약 인트론과 엑손이 이렇게 연결되어 있는 유전자가 있다고 가정하자. - 는 인트론이며, 숫자는 엑손이다.
프로모터-1-----2---3-----4----5-종결부위
그러면 이어맞추기를 이용하여 12345 라는 RNA를 만들 수도 있으나, 대체 이어맞추기를 이용하며 엑손의 일부를 누락시킬 수도 있다.
즉, 엑손 3이 인트론과 함께 제거되며 1245 라는 RNA가 만들어 질 수도 있으며, 엑손 4가 제거되어 1235 라는 RNA가 만들어 질 수도 있다.
그러면 하나의 유전자에서 엄청나게 많은 종류의 단백질이 만들어 질 수 있겠군요? 그렇다.
이것들을 RNA의 이성질체(Isoform)라 부르며 대략 인간 유전자의 75% 정도가 대체 이어맞추기에 의해 유전자당 하나 이상의 이성질체를 만드는 것으로 추정된다.
하지만 이것을 조절하기 위해 상당히 복잡한 기전들이 사용된다. 대체 이어맞추기에 대해선 따로 긴 분량을 투자하여 설명하게 될 것이다.
그런 고로, 여기서는 기본적인 이어맞추기 기전에 대해 살펴본다.
우선 짚고 가야 하는 사실은, 무의미한 것처럼 늘어서 있는 염기서열 가운데에서 어떻게 인트론 부위와 엑손 부위를 구분하는가이다.
Boundary sequence
우선적으로 생각해 볼 수 있는 것은, 경계를 형성하는 특정한 염기서열(Boundary sequence)이 있어, 이것을 인식하는 방법일 것이다.
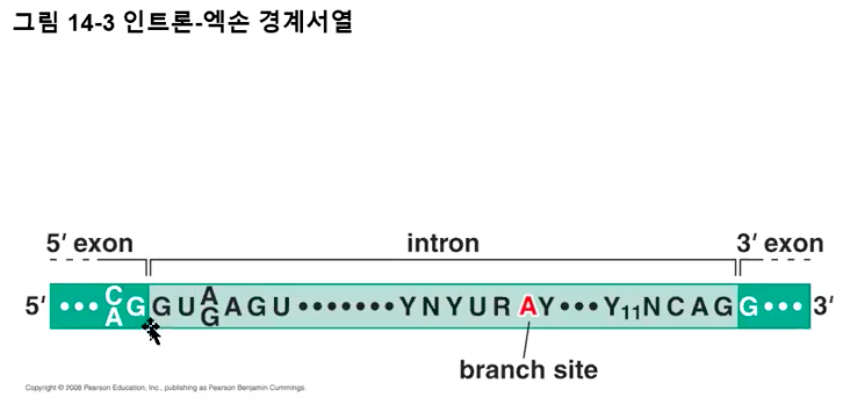
엑손과 인트론의 경계에 위치한 서열을 이어맞추기 자리(Splice site)라 부르는데, 인트론의 5' 말단 경계를 5' 이어맞추기 자리라 하고, 인트론의 3' 말단 경계를 3' 이어맞추기 자리라고 한다.
그리고 이어맞추기에 필요한 매우 중요한 부위가 있는데, 인트론의 3' 말단 가까이에 위치한 분기점 자리(BS, Branch point site)가 있다.

위 모식도를 보면, pre-mRNA에서 5' 이어맞추기 자리와 분기점 자리, 3' 이어맞추기 자리가 순서대로 나타나 있다.
그런데 염기 서열을 보면 듣도 보도 못한 R, Y, N 이라는 서열이 보일 것이다. R은 퓨린 염기, Y는 피리미딘 염기, N은 어떤 염기든 들어갈 수 있다는 것을 뜻한다.
여기에서 특히 중요한 것이 5' 이어맞추기 자리의 GU와, 3' 이어맞추기 자리의 AG다. 모든 진핵생물에 있어 이 부분이 가장 잘 보존되어 있으며, 이 부위가 손상될 경우 이어맞추기가 일어날 수 없다.
그런데 서열이 너무 단순한 것 아닌가요? 그렇다. 하지만 인트론에 결합하는 수많은 인트론 결합 단백질들이 이어맞추기를 조절하므로 부정확한 위치에서의 이어맞추기가 일어나는 것을 막는다.
인트론 결합 단백질은 또한 일종의 마커로써 기능할 수도 있어서 인트론을 갖는 mRNA가 세포질로 나가지 못 하도록 하는 기능도 한다.
어쨌거나, 5', 3' 이어맞추기 자리와 함께 중요한 것이 바로 분기점 자리다.
분기점 자리는 3' 말단에 가까운 곳에 위치한 아데노신(A)인데, 그 뒤로 피리미딘 연속서열(PPT, Polypyrimidine tract)이 나타난다.
인트론에는 수많은 아데노신이 있지만, 아데노신 뒤에 피리미딘 연속서열이 나타날 경우 그 아데노신이 바로 분기점 자리가 되는 것이다.
그러면 이제 어떻게 이어맞추기가 일어나는지를 살펴보도록 하자.

우선, 이어맞추기는 두 번의 연속적인 에스테르 교환(Transesterification) 반응에 의해 인트론이 제거되는 과정이다.
에스테르 교환이라고 해서 어렵게 생각할 것은 없고, 리보뉴클레오티드를 연결하고 있는 결합이 인산디에스테르(Phosphodiester) 결합인데, 이 결합을 두 번 자르고, 두 번 붙인다는 말이다.
위의 모식도를 보면, 우선 분기점 자리의 아데노신에 의해 5' 말단 이어맞추기 자리의 절단이 일어남과 동시에 아데노신의 2번째 탄소자리에서 인산디에스테르결합이 일어난다.
어 그게 가능한가요? RNA를 이루는 리보뉴클레오시드인 아데노신에는 2번째 탄소에도 -OH가 있다는 것을 기억하자.
그러고 나면, 엑손의 '3 말단에 노출된 -OH가 인트론의 3' 말단 이어맞추기 부위를 절단하는 동시에 엑손끼리의 연결이 일어날 수 있다.
그렇게 되면 남은 인트론은 고리 형태로 제거될 수 있다.
이것과 반응 자체는 동일한데 서로 분리된 두 RNA 사슬에 있던 두 엑손이 단일 mRNA로 이어맞추기가 되는 경우가 있다. 비교적 특이한 경우로, 이 경우를 트랜스-이어맞추기(Trans-splicing)라 부른다.

다음과 같이 서로 분리된 RNA의 두 엑손이 하나의 mRNA로 결합하면서 고리형이 아닌 Y자 형의 인트론 절편이 생겨난다는 점이 다르지만 반응 자체는 동일하다.
본래 이 반응은 ATP를 소모하지 않는 반응이지만, 이어맞추기에 사용되는 여러 효소들이 제대로 작동하기 위해서 실제로는 많은 양의 ATP를 소모한다.
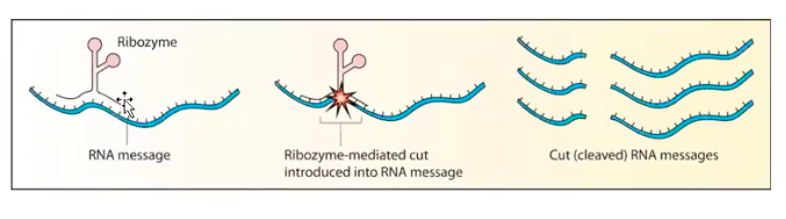
Ribozyme
이어맞추기에 관여하는 효소는 여러 종류가 있는데, 대부분이 RNA 효소인 리보자임(Ribozyme)이다.
여기에 관여하는 인자들은 매우 정교하고 유기적으로 작동하는데, 흔히 인자들을 모두 묶어 이어맞추기 복합체(Spliceosome)라 부른다.
이어맞추기 복합체에는 주요 이어맞추기 복합체(Major spliceosome)과 부 이어맞추기 복합체(Minor spliceosome)이 있는데, 여기선 주요 이어맞추기 복합체에 대해 설명한다.
부 이어맞추기 복합체는 포스트의 끝에서 매우 짧게 설명하겠다. 사실상 주요 이어맞추기 복합체와 거의 차이가 없다.
Major spliceosome
주요 이어맞추기 복합체에는 보통 5개의 RNA와 150개 가량의 단백질로 구성되는데, 여기에 이용되는 RNA들을 통칭하여 snRNA(Small nuclear RNA)라 부른다.
snRNA에는 U1, U2, U4, U5, U6이 있다. 왜 3번이 없는지는 나도 모른다.
이들은 대략 100~300b으로 이루어진 짧은 RNA들인데, 이들 각각이 여러개의 단백질과 결합하여 리보자임을 형성한 것을 snRNP(Small nuclear ribonuclear protein)라 부른다.
각각의 snRNP는 이어맞추기에 필요한 세 가지 역할을 수행하는데, 우선 5' 이어맞추기 자리와 분기점 자리를 인지하여 이 둘을 서로 가까이 위치하게 해 RNA의 절단과 결합을 촉매한다.
효소로 리보자임이 필요한 것 역시 지정된 형태의 특정 염기서열을 정확하게 인식하기 위해서다. 상당히 서열 특이적이다.
하지만 이어맞추기 복합체에 이들 리보자임만 존재하는 것은 아니고 RNA에 결합하지 않는 몇몇 단백질도 반응에 참가한다. 역할은 대부분 보조하는 역할이다.
몇 가지 예를 들자면, U2AF(U2 anxilliary factor)는 피리미딘 연속서열과 3' 이어맞추기 자리를 인지하여 BBP(Branch-point binding protein)가 분기점 자리에 결합하는 것을 돕는다.
BBP가 분기점 자리에 결합하면, U2 snRNP가 이를 인식하고 BBP와 교체되어 결합한다.
그리고 다른 snRNP들이 올바른 위치에 위치하도록 보조하는 RNA 융합인자(RNA-annealing factor)나, snRNA와 mRNA 사이의 결합을 분리하기 위한 DEAD 박스 헬리카아제(DEAD-box helicase) 등이 존재한다.

가장 일반적으로 많이 일어나는 mRNA의 이어맞추기는 다음과 같은 과정을 통해 일어난다.
차례대로 살펴보도록 하자. 우선, 5' 이어맞추기 자리를 U1 snRNP가 인지하여 결합하고, 피리미딘 연속서열을 U2AF 단백질이 인식하여 결합한다.
BBP 단백질은 U2AF에 의해 분기점 자리로 유도되는데 이 단계를 초기 복합체(E complex, Early complex)라 부른다.
뒤이어 BBP를 인식하여 U2 snRNP가 그 BBP를 대체하여 분기점 자리에 결합한다. 이 과정에서 ATP가 소모된다.
이 단계에서 재미있게도 분기점 자리의 A가 결합을 하지 못 하고 벌지(Bulge)를 형성한다. 툭 튀어나온다는 말이다.
이 A는 염기쌍을 형성하지 않으므로 5' 이어맞추기 자리를 공격할 수 있게 된다. 이 단계를 A 복합체(A complex)라 부른다.
그러면 뒤이어 U4~U6 snRNP가 tri-snRNP의 형태로 복합체에 참여하여 B 복합체로 전환되며, 인트론이 고리 형태를 이루는 구조적 변형이 일어난다.
U1과 U6이 충분히 가까워지면, U6은 U1을 대체하여 5' 이어맞추기 자리에 결합하고, U6과 U2 사이에 위치하던 U4가 빠져나가며 U6과 U2의 상호작용이 일어날 수 있게 된다.
U6은 '5 이어맞추기 자리와 결합한 상태이며, U2는 분기점 자리에 결합한 상태다. 이 상태에서 다른 방향에서 두 리보자임의 결합이 일어남에 따라 5' 이어맞추기 자리와 분기점 자리 역시 매우 근접할 수 있다.
이 상태가 C 복합체(C complex)이며, U6과 U2의 상호작용에 의해 활성부위가 형성될 수 있다.
그 결과 분기점 자리에 벌지를 형성하고 있는 A에 의해 첫번째 에스테르 교환(Transesterification)이 일어난다.
5'와 3' 이어맞추기 자리에서 일어나는 두 번째 에스테르 교환은 첫번째 에스테르 교환이 일어난 이후, U5가 3' 이어맞추기 자리를 인지하여 두 엑손을 나란히 위치시킴으로써 일어난다.
성공적으로 이어맞추기가 완료되면 이어맞추기 복합체는 인트론과 함께 떨어져 나가는데, snRNP들은 할 일을 마친 후 바로 인트론에서 떨어져 나오지 않고 인트론이 분해된 후에 떨어져 나온다.
자, 이 쯤에서 의문이 생길 수 있을 것이다. 5'과 3' 이어맞추기 자리는 상당히 짧은 염기서열로만 이루어져 있다.
물론 U1과 U2가 염기 특이적으로 인식하므로 제법 엄격하게 인지를 하게 되지만 그래도 여전히 쉬운 문제가 아니다.
만약 pre-mRNA 분자를 시퀀싱해서 염기서열을 쭉 쓴 후에 엑손과 인트론을 찾아보라면 찾을 수 있겠는가?
그런데 이어맞추기 복합체는 어떻게 이 자리를 알아서 정확하게 이어맞추기를 하는가?
이어맞추기에는 흔히 두 종류의 실수가 일어날 수 있는데, 첫번째로는 엑손을 중간에 하나 건너뛰고 이어맞추기를 하는 경우다.
물론 대체 이어맞추기(Alternative splicing)에서는 이런 경우가 일어나지만, 의도하지 않았는데 일어나게 되면 문제가 된다.
다음으로, 본래 인지되어야 할 이어맞추기 자리가 아니라 엉뚱한 위치에 비슷한 유사서열을 대신 인식하는 경우다.
이런 경우도 이어맞추기가 엉뚱하게 이루어지므로 제대로 단백질이 발현되지 않는다.
그래서 RNA 전사 과정에서 RNA 중합효소의 C 말단 꼬리에 이어맞추기에 관련된 여러 인자가 결합하고 있다가 엑손에서 다음 엑손까지 전사하고 나면 즉시 이어맞추기를 수행하는 것이다.
그러면 최소한 중간에 위치한 엑손을 거르고 이어맞추기를 하는 경우는 줄어들게 될 것이다.
그리고 이것과 함께 사용되는 다른 기작으로써 SR 단백질과 같은 엑손 마커(Marker) 단백질을 이용하는 방법이다.
SR 단백질은 세린과 아르기닌이 매우 풍부한 일련의 여러 단백질들을 통틀어 일컫는 말인데, 엑손 내의 ESE(Exonic splicing enhancer)라는 서열에 결합한다.
이 부위에 결합한 SR 단백질은 이어맞추기에 관련된 여러 인자들과 상호작용하여 근처로 모으고, 엑손에 있는 적절한 이어맞추기 자리에 위치하도록 유도한다.
인트론이 엑손에 비해 훨씬 더 길다는 사실을 생각한다면, 엑손이 있는 곳으로 이어맞추기 인자를 끌어모으는것 만으로도 이어맞추기의 정확도를 크게 향상시킬 수 있을 것이다.
특히 SR 단백질은 U2AF 단백질을 3' 이어맞추기 자리가 있는 곳으로 모으고, U1 snRNP는 5' 이어맞추기 자리로 모아 이어맞추기의 정확성을 높인다.
(분기점 자리와 피리미딘 연속서열은 3' 이어맞추기 자리 근처에 위치한다는 것을 잊지 말자)
또 한, SR 단백질은 이 뿐만 아니라 나중에 설명하게 될 대체 이어맞추기를 조절하는 단백질이기도 하다.
여기까지가 주요 이어맞추기 복합체에 대한 설명이다. 부 이어맞추기 복합체는 정말 빠르게 간단하게 끝내도록 하겠다.
부 이어맞추기 복합체는 별개의 이어맞추기 서열을 인식하는데, 주요 이어맞추기 복합체가 인식하는 서열이 GU-AG인 것에 반해, 부 이어맞추기 복합체가 인식하는 서열은 AU-AC 서열이다.
(5', 3' 이어맞추기 자리를 말하는 것이다. 분기점 자리도 약간의 서열 차이는 있지만 동일하게 A와 피리미딘 연속서열을 갖는다)
당연히 참가하는 snRNP들 역시 다르다. 하지만 기전 자체는 완전히 동일하다. 굳이 또 설명할 필요는 없으니 비교도만 보자.

Group l , Group ll self-splicing
앞에서는 이어맞추기 복합체(Spliceosome)에 의해 이어맞추기가 일어나는 과정에 대해 살펴보았다.
그러나 어떤 인트론들은 이어맞추기 복합체가 없어도 스스로 이어맞추기를 할 수 있다.
이런 인트론들을 자가 이어맞추기 인트론(Self-splicing intron)이라 부르는데 Ⅰ군과 Ⅱ군의 자가 이어맞추기 인트론으로 구분할 수 있다.

어떤 차이를 갖는가? 참고로 저 둘 모두 RNA가 스스로 알아서 이어맞추기를 한다는 공통점이 있다.
Group ll self-splicing
우선, 오른쪽의 Ⅱ군 자가 이어맞추기 인트론을 먼저 보자. 이 인트론은 이어맞추기 방식 자체도 이어맞추기 복합체를 이용한 이어맞추기 방식과 동일하다.
즉, 분기점 자리(Branch point site)에 위치한 A가 벌지(Bulge)를 형성하여, 2번째 탄소가 갖고 있는 -OH기를 이용해 5' 이어맞추기 자리를 공격하여 고리형 인트론을 형성한다.
Group l self-splicing
그러나 Ⅰ군 자가 이어맞추기 인트론은 조금 다른 것 같다. 우선 분기점 자리가 A가 아니라 G인데, 떨어져 나온 인트론이 고리형이 아니라 선형이다.
거기에 자세히 보니 분기점 자리의 G는 RNA에 포함되어 있지 않은 독립된 리보뉴클레오티드다! 이 녀석이 상보적으로 RNA와 달랑 혼자서 결합하고 있는 걸까?
그것은 아니다. 재미있게도, Ⅱ군 자가 이어맞추기 인트론에서 분기점 자리가 벌지를 형성하는 것과는 달리, Ⅰ군 자가 이어맞추기 인트론에서는 분기점 자리가 포켓(Pocket)을 형성한다.
그 포켓 안에 독립된 G-리보뉴클레오티드가 끼워져 있는 형태다. 그래서 A 대신에 G를 이용하여 이어맞추기를 진행한다.
그런데 어떻게 이들은 이어맞추기 복합체의 도움 없이 스스로 알아서 이어맞추기를 진행할 수 있는가? 그것은 인트론 서열의 특이성 때문이다.
저~~~~ 앞에서 RNA의 성질에 대해 설명할 때 RNA의 가장 두드러지는 특징은 어떤 것이 있다고 했던가?
RNA는 스스로 접히고 꼬여서 독자적인 3차적인 구조를 형성할 수 있고, 그것이 효소로써 작용할 수도 있다고 했었다.
뒤이어 망치머리 리보자임(Hammerhead ribozyme)이 작동하는 방식에 대해서도 설명한 바 있을 것이다.
망치머리 리보자임은 별다른 효소의 도움 없이도 스스로 자신을 분해하는 RNA다. 여기서 감이 잡히는가?

위 모식도는 이어맞추기 복합체가 이어맞추기를 위해 형성하는 구조(a)와, Ⅱ군 자가 이어맞추기 인트론이 형성하는 구조(b)를 나타낸다.
놀라울 만큼 닮았다고 생각되지 않은가? 즉, 인트론 자체가 snRNP의 기능을 모두 수행하는 것이다.
Ⅰ군 자가 이어맞추기 인트론 역시 마찬가지 원리를 갖는다. 인트론 자체가 저렇게 꼬이고 접히는데, 대신 복제 분기점에서 A가 아니라 독입된 G-리보뉴클레오티드를 사용한다는 점만 다르다.
이러한 자가 이어맞추기 인트론의 효소 활성이 초창기의 생물체들이 유전 물질로써 자가 복제-분리될 수 있는 RNA를 이용하였다는 주장에 힘을 실어주는 하나의 근거가 될 수 있다.
즉, 초기에는 RNA가 효소의 역할을 담당했으나 차츰 단백질로 그 기능이 옮겨갔다는 것이다.
'🧬 Bio > 분자생물학' 카테고리의 다른 글
| 분자생물학 (15) - Translation (1) | 2020.12.03 |
|---|---|
| 분자생물학 (14) - Alternative splicing, Exon shuffling, RNA editing (0) | 2020.11.30 |
| 분자생물학 (13) - 진핵세포의 전사 (0) | 2020.11.26 |
| 분자생물학 (13) - 전사기작 (1) | 2020.11.25 |
| 분자생물학 10장 (2) - DNA 손상 (0) | 2020.10.15 |